|
Applications
Resistance Gene Enrichment Sequencing(RenSeq) |
|
抵抗性遺伝子濃縮シーケンシング(RenSeq:Resistance gene enrichment sequencing)のワークフローにより、農作物のゲノム内にある複雑な抵抗性遺伝子(R遺伝子)ファミリーの包括的な研究が可能になります(Jupe ea.al. 2013)。R遺伝子は通常、ヌクレオチド結合「NB」および、ロイシンリッチリピート「LRR」の2種類のドメインを持つため、「NB-LRR」または「NLR」構造タンパク質として知られます(Jupe. et.al 2013)。これらの遺伝子は多種多様な病原体、真菌や卵菌、細菌、昆虫、ウイルスなどから保護するように機能するため重要です(Schulze-Lefert & Panstruga, 2011)。したがって、病害抵抗性遺伝子は農業の観点から理解することが重要であり、繁殖プログラムや関連する研究活動で最大限の有用性を発揮するために利用できます。
RenSeqはR遺伝子の濃縮を提供しますが、特定の病原体に対する個々の遺伝子の防御機構の役割を理解することがこの発見プロセスの究極の目標です。AgRenSeq (
Associated
genetics
R gene
enrichment
sequencing)はR遺伝子の役割を特定するための次の進歩です。R遺伝子内の耐病性形質と高特異性免疫受容体ドメインの間の相関関係を提供します。
下記のJohn Innes Centreのビデオでは、野生および古代の品種のスクリーニングがさまざまな病原体に対する抵抗性をもたらすDNAドメインをどのように明らかにするかを説明しています。
RenSeqアプリケーションは、R遺伝子とその機能を確認し、これらの遺伝子の発現をオンにする病原体自体を特定するための、新しい独自の手法にまで拡大し続けています。
R遺伝子の同定と分析の簡潔なタイムライン:
- MutRenSeq

クローニング前のR遺伝子の突然変異誘発(Mutagenesis)とターゲットシーケンスによる同定
- PenSeq

病原性をコードする遺伝子を同定するための病原体毒性遺伝子(Pathogen virulent gene)のキャプチャとシーケンス
- AgRenSeq

形質と遺伝子の間の相互作用を同定するためのアソシエーション遺伝学(Association genetics)RenSeq
- dRenSeq

作物中の既知のR遺伝子の信頼性の高い同定と完全な配列検証のための診断(Diagnostic)RenSeq
作物のゲノムには通常、数百以上の異なるNLR領域があり、それぞれの長さは数Kbです。 これらの領域は独立している場合もあれば、他のNLR領域に近いクラスターにある場合もあります(Jupe et al 2013)。メンバー間の配列類似性が比較的高く、大きく複雑な遺伝子ファミリーの場合、ローパス全ゲノムシーケンスまたはアンプリコンベースのシーケンスなどの特定シーケンス法において課題を生じます。一方でハイブリダイゼーションキャプチャ法は深さと幅の両方の観点から、堅牢なターゲット回復の理想的なバランスを提供します。
ハイブリダイゼーションでは、NGSライブラリは変性され、図1に示されるように関心領域に相補的なカスタマイズされたビオチン化プローブがハイブリダイズします。Daicel Arbor Biosciences社などのベンダーが提供する手頃なオリゴ合成技術により、数万の固有のプローブを含むカスタムプローブセットを生成できます。これにより、1回のRenSeq実験で特定のゲノム内の既知のR遺伝子の完全な配列多様性を含めることができます。このアプローチは既知のNLR領域と新規の領域の回復を実現します。ハイブリダイゼーションベースのキャプチャの性質により、特定のプローブから最大20%までの配列分岐を持つ分子を濃縮します。このように濃縮されるR遺伝子は、特定のゲノム配列のアノテーション中に欠落しているか、リファレンスゲノムから完全に欠落している可能性があります。したがって、ハイブリダイゼーションキャプチャは新規のR遺伝子ゲノムコンテンツを見つけるためのツールとして機能するとともに、ゲノム中のNLR領域のアノテーションを改善するツールにもなりえます(Jupe et.al. 2013)。
1つまたは複数の完全なNLR遺伝子(通常は長さ3.2 kb)を完全にシーケンスする機能により、R遺伝子研究に関連するゲノム領域の高い解像度が得られます。
myBaits®カスタムプローブライブラリを使用したハイブリダイゼーションキャプチャの利点は、任意のシーケンスプラットフォームと完全に互換性があることです。つまり、同じプローブセットを使用して、イルミナ
®ショートリードシーケンスまたはロングリードシーケンスプラットフォーム、OxfordNanopore
®やPacBio
®用に調製されたDNAサンプルを濃縮できます。Daicel Arbor Biosciences社は、RenSeqプロジェクトについてのご相談や、カスタムパネルの設計をサポートします。また、
myReads®NGSサービスチームの科学者によるフルサービスプロジェクトの作成を喜んでお手伝いします。
|
|
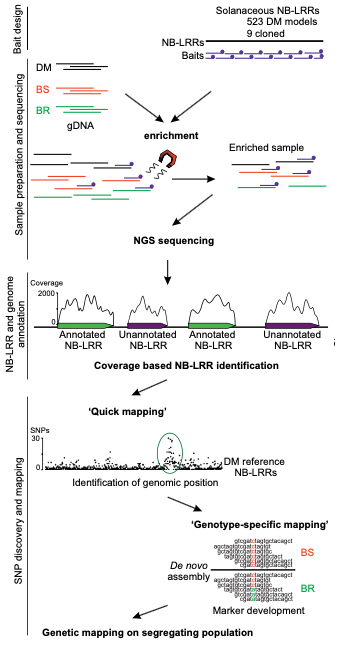
図1. カスタマイズされたNB-LRR RNA Bait-libraryを使用して、ゲノムDNAからNB-LRR配列を濃縮します。
標準のイルミナシーケンスとBWAマッピングにより、追加のNB-LRR遺伝子座の識別が可能になりました。病原体耐性を分離するマーカーを迅速に特定するために、分離集団の中で最も耐性のある表現型植物(BR)および最も感受性の高い表現型植物(BS)のバルクゲノムDNAを同じワークフローにかけました。次に、イルミナのリードを「クイックマッピング」分析に適用して、予測されたNB-LRRにマッピングし、遺伝子ごとのBR固有のSNPの数をプロットすることで、おおよその染色体位置を特定しました。NB-LRR濃縮リードのdenovoアセンブリは、BR固有のSNPを呼び出すための遺伝子型固有の参照として使用されます。 Jupe et. al. 2013の画像提供。 |
以下は、耐病性遺伝子の研究の中でDaicel Arbor Biosciences社
myBaits® カスタムキット が利用された論文です。
過去数年に渡り、オリジナルのRenSeqアプローチから多くのバリエーションが発明されおり、その中から選択されています。
Bettgenhaeuser, Hernandez-Pinzon, et al. 2021.
The barley immune receptor Mla recognizes multiple pathogens and contributes to host range dynamics.

Nature Communications, 12(1), pp.1-14.
Upadhyaya, Mago, et al. 2021.
Genomics accelerated isolation of a new stem rust avirulence gene-wheat resistance gene pair.

Nature Plants, 7(9), pp.1220-1228.
Seong, Seo, et al. 2020.
Evolution of NLR resistance genes with noncanonical N‐terminal domains in wild tomato species.

New Phytologist, 227(5), pp.1530-1543.
Narang et al. 2020.
Discovery and characterisation of a new leaf rust resistance gene introgressed in wheat from wild wheat Aegilops peregrina.

Scientific Reports, 10(1), pp.1-9.
Lin et al. 2020.
RLP/K enrichment sequencing; a novel method to identify receptor‐like protein (RLP) and receptor‐like kinase (RLK) genes.

New Phytologist, 227(4), pp.1264-1276.
Armstrong et al. 2019.
Tracking disease resistance deployment in potato breeding by enrichment sequencing.

Plant Biotechnology Journal
Arora et al. 2019.
Resistance gene cloning from a wild crop relative by sequence capture and association genetics.

Nature Biotechnology
Cevik et al. 2019.
Transgressive segregation reveals mechanisms of Arabidopsis immunity to Brassica-infecting races of white rust (Albugo candida).

PNAS
Grech-Baran et al. 2019.
Extreme resistance to Potato Virus Y in potato caaryying the Ry(sto) gene is mediated by a TIR-NLR immune receptor.

Plant Biotechnology Journal
Jouanin et al. 2019.
Development of GlutEnSeq capture system for sequencing gluten gene families in hexaploid bread wheat with deletions or mutation induced by gamma-irradiation or CRISPR/Cas9.

Journal of Cereal Science
Narang et al. 2019.
Fine mapping of Aegilops peregrina co-segregating leaf and stripe rust resistance genes to distal-most end of 5DS.

Theoretical and Applied Genetics
Van de Weyer et al 2019.
A Species-Wide Inventory of NLR Genes and Alleles in Arabidopsis thaliana.

Cell
Brabham et al. 2018.
An ancient integration in a plant NLR is maintained as a trans-species polymorphism.

bioRxiv
Giolai et al. 2018.
Targeted capture and sequencing of gene-sized DNA molecules.

Biotechniques
Marchal et al. 2018.
BED-domain-containing immune receptors confer diverse resistance spectra to yellow rust.

Nature Plants
Thilliez et al. 2018.
Pathogen enrichment sequencing (PenSeq) enables population genomic studies in oomycetes.

New Phytologist
Giolai et al. 2017.
Comparative analysis of targeted long read sequencing approaches for characterization of a plant's immune receptor repertoire.

BMC Genomics
Witik et al. 2016.
Accelerated cloning of a potato late blight-resistance gene using RenSeq and SMRT sequencing.

Nature Biotechnology
Steuernagel et al. 2016.
Rapid cloning of disease-resistance genes in plants using mutagenesis and sequence capture.

Nature Biotechnology