myBaitsターゲットエンリッチメントを含むNGSサービス
myReadsは、ハイブリダイゼーションベースのターゲットキャプチャキット(myBaits)を利用した次世代シーケンスサービスです。お客様よりご提供いただいたDNAまたはRNAをmyReadsチームが受け取った後、DNA/RNAライブラリ調製および、ターゲットキャプチャ、次世代シーケンスを行い、データをご返却します。
日頃より、さまざまなプロジェクトのプローブのカスタム設計やターゲットエンリッチメント、ターゲットシーケンスを行う経験豊富なmyReadsのサイエンスチームがあらゆる生物種、あらゆるアプリケーションのシーケンスサービスをお届けします。
特長
- 実績のあるシステム
myBaitsを利用した高度なライブラリー調製により一貫した結果を提供します。 - あらゆるサンプルタイプ
新鮮なDNAから分解されたDNA、メタゲノミクスや古代DNAまで利用可能です。 - 共同プロセス
お客様の研究プロジェクトや目的に合わせたNGSサービス全体およびカスタムキットのコンサルティングを行います。 - 複数のシーケンスプラットフォーム:
Element Biosciences®, Illumina®, PacBio®の複数のシーケンスプラットフォームからご選択いただけます。 - ボリュームディスカウント
大規模なプロジェクトでは、段階的な価格を提供します。
| 
|
| ターゲットキャプチャキット myBaits
プローブとターゲットの配列が完全に一致している必要はなく、
20%強の違いを許容し、エンリッチメントが可能です。
|
|
| |
ページの先頭に戻る
サービス概要
myReads カスタムNGSサービスは、お客様より提供されたDNAまたはRNAをmyReadsサイエンスチームが受け取った後、ライブラリ調製、ハイブリダイゼーションベースのターゲットキャプチャ、シーケンスを行い、データをお返しするサービスです。
ターゲットキャプチャのステップでは、標的の濃縮と非標的の洗浄を行うため、シーケンスの効率を高め、コストを抑えたターゲットシーケンスを行えます。
| NGSライブラリ調製 |
| ターゲットエンリッチメント
(myBaits) |
| ショートリード、
ロングリードシーケンス |
- ショートインサートのライブラリ調製(DNA/RNA)
- ロングインサートのライブラリ調製(DNA)
- 劣化・分解のある古代DNA用の特別な二本鎖または一本鎖ライブラリ調製
| - myBaits 新しいカスタムパネル(無償設計サポート)
- myBaits, Expert, 特定のアプリケーション用の既存パネル
| - Element Biosciences® AVITI
- Illumina® NovaSeq
- Illumina® MiSeq
- PacBio® Revio
- PacBio® Sequel II
- その他
|
|
|
|
ページの先頭に戻る
サービスの流れ
| 1. お客様のプロジェクト内容のヒアリングと見積もり |
| 2. (カスタムの場合) myBaitsカスタムハイブリダイゼーションキャプチャキットの設計 |
| 3. ガイドラインに沿ったサンプルの調製とDNA又はRNAの出荷 |
| 4. デジタルNGS FASTQ リードデータのご返却 |
|
| 
myReads
初回お伺い項目 |
| 
myReads
サンプル調製ガイド |
|
お問い合わせは【myReads初回お伺い項目】にご記入のうえ、
reagents@primetech.co.jpまでご連絡ください。
ページの先頭に戻る
NGSサービスパッケージ
詳細な条件は
Service Policies 
をご確認ください。
- Custom Captureパッケージ
| Standard DNA | 新鮮なサンプルより抽出された高品質のDNA |
| サンプル要件 | 高分子量 10 kbp以上、純度 UV260/280 1.7~1.9、DNA量 500 ng以上 |
| サービスに含まれる内容 | - QC、フラグメンテーション、ライブラリ調製、8-12plexでのキャプチャ、AVITIでの1サンプルあたり1Gbp、PE150のシーケンシング
- 要件を満たすサンプルのライブラリ調製のパフォーマンスコミットメント
- QCレポートより、どのサンプルを残すか、廃棄するか、追加するかを決定することができます
|
| Degraded DNA | 古い標本や植物標本からのDNA、品質不明のDNA |
| サンプル要件 | さまざまな分子量 、さまざまな純度(UV260/280値)、DNA量 10 ng以上 |
| サービスに含まれる内容 | - 拡張QC、フラグメンテーション(必要に応じ)、ライブラリ調製(必要に応じ特殊なケミストリーを用いる)、4-6plexでのキャプチャ、AVITIでの1サンプルあたり1Gbp、PE150のシーケンシーング
- 劣化したサンプルのパフォーマンスは保証されません
- QCレポートより、どのサンプルを残すか、廃棄するか、追加するかを決定することができます
|
| Ancient DNA double-stranded | 博物館や歴史館などからの古代DNA |
| サンプル要件 | 低分子量 1 kbp以下 |
| サービスに含まれる内容 | - QC、特別なケミストリーを利用した二本鎖ライブラリ調製(古代DNAのための「ゴールドスタンダード」法)、2-4plexでのdual-roundキャプチャ、AVITIでの1サンプル当たり1Gbp、PE150のシーケンシング
- 古代サンプルのパフォーマンスは保証されません
- QCレポートより、どのサンプルを残すか、廃棄するか、追加するかを決定することができます
|
| Ancient DNA Single-stranded | 博物館などの古代DNA、またはcfDNA(cell-free DNA)、または一本鎖cDNA |
| サンプル要件 | 低分子量 1 kbp以下 |
| サービスに含まれる内容 | - QC、特別なケミストリーを利用した一本鎖ライブラリ調製、2-4plexでのdual-roundキャプチャ、AVITIでの1サンプル当たり1Gbp、PE150のシーケンシング
- 古代サンプルのパフォーマンスは保証されません
- QCレポートより、どのサンプルを残すか、廃棄するか、追加するかを決定することができます
|
| Long-insert DNA | PacBioシーケンシングを目的としたDNAまたはcDNA用 |
| サンプル要件 | 高分子量 10 kbp以上、純度 UV260/280 1.7~1.9、DNA量 5 μg以上 |
| サービスに含まれる内容 | - QC、キャプチャのためのロングインサートライブラリ調製、3-plexでのキャプチャ、PacBio Revio cellあたり24サンプルのSMRTbell調製
- 要件を満たすサンプルのライブラリ調製のパフォーマンスコミットメント
- QCレポートより、どのサンプルを残すか、廃棄するか、追加するかを決定することができます
|
| Standard RNA | 高品質のRNAまたはmRNA |
| サンプル要件 | 純度 UV 260:280 1.9~2.1、RNA量 250 ng以上 |
| サービスに含まれる内容 | - QC、DNase処理、精製、8-12plexでのキャプチャ、AVITIでの1サンプル当たり1Gbp、PE150のシーケンシング
- ヒト・マウス・ラットサンプルでは、mRNAキャプチャとrRNA除去のオプション
- 要件を満たすサンプルのライブラリ調製のパフォーマンスコミットメント
- QCレポートより、どのサンプルを残すか、廃棄するか、追加するかを決定することができます
|
▲ リストへ戻る
- Expert Capture パッケージ
下記リストのmyBaits Expert panelをご選択の場合、さらにStandard DNAのサンプル要件を満たす場合ご利用いただけます。
分解DNAの場合、追加料金でご対応いたします。
| Ultraconserved Elements | 超保存エレメント用ターゲットキャプチャキット myBaits Expert UCEよりパネルを選択 |
| サンプル要件 | 高分子量 10 kbp以上、純度 UV260/280 1.7~1.9、DNA量 500 ng以上 |
| サービスに含まれる内容 | - QC、フラグメンテーション、ライブラリ調製、マルチプレックスでのキャプチャ、AVITIでの1サンプルあたり0.6 Gbp、PE150のシーケンシング
- 要件を満たすサンプルのライブラリ調製のパフォーマンスコミットメント
- QCレポートより、どのサンプルを残すか、廃棄するか、追加するかを決定することができます
|
| Angiosperms-353 | 被子植物オーソログ用ターゲットキャプチャキット myBaits Expert Angiosperms 353 |
| サンプル要件 | 高分子量 10 kbp以上、純度 UV260/280 1.7~1.9、DNA量 500 ng以上 |
| サービスに含まれる内容 | - QC、フラグメンテーション、ライブラリ調製、マルチプレックスでのキャプチャ、AVITIでの1サンプルあたり0.6 Gbp、PE150のシーケンシング
- 要件を満たすサンプルのライブラリ調製のパフォーマンスコミットメント
- QCレポートより、どのサンプルを残すか、廃棄するか、追加するかを決定することができます
|
| 16S-Hyb | 細菌ゲノム用ターゲットキャプチャキット myBaits Expert 16S-Hyb |
| サンプル要件 | 高分子量 10 kbp以上、純度 UV260/280 1.7~1.9、DNA量 500 ng以上 |
| サービスに含まれる内容 | - QC、フラグメンテーション、ライブラリ調製、マルチプレックスでのキャプチャ、AVITIでの1サンプルあたり3 Gbp、PE150のシーケンシング
- 要件を満たすサンプルのライブラリ調製のパフォーマンスコミットメント
- QCレポートより、どのサンプルを残すか、廃棄するか、追加するかを決定することができます
|
| Respiratory Viruses - DNA | 呼吸器系ウイルス用ターゲットキャプチャキット myBaits Expert Respiratory Virus |
| サンプル要件 | 高分子量 10 kbp以上、純度 UV260/280 1.7~1.9、DNA量 500 ng以上、一本鎖あるいは二本鎖DNA |
| サービスに含まれる内容 | - QC、フラグメンテーション、一本鎖あるいは二本鎖用のライブラリ調製、マルチプレックスでのキャプチャ、AVITIでの1サンプルあたり1 Gbp、PE150のシーケンシング
- 要件を満たすサンプルのライブラリ調製のパフォーマンスコミットメント
- QCレポートより、どのサンプルを残すか、廃棄するか、追加するかを決定することができます
- 一本鎖DNAはRespiratory Viruses - RNAと組み合わせることも可能です。
|
| Respiratory Viruses - RNA | 呼吸器系ウイルス用ターゲットキャプチャキット myBaits Expert Respiratory Virus |
| サンプル要件 | 純度 UV260/280 1.7~2.1、RNA量 250 ng以上 |
| サービスに含まれる内容 | - QC、DNase処理、精製、フラグメンテーション、directionalライブラリ調製、マルチプレックスでのキャプチャ、AVITIでの1サンプルあたり0.6 Gbp、PE150のシーケンシング
- ヒト・マウス・ラットサンプルでは、mRNAキャプチャとrRNA除去のオプション
- 要件を満たすサンプルのライブラリ調製のパフォーマンスコミットメント
- QCレポートより、どのサンプルを残すか、廃棄するか、追加するかを決定することができます
- Respiratory Viruses - DNA (一本鎖)と組み合わせることも可能です。
|
| Wheat Exome | コムギエクソーム用ターゲットキャプチャキット myBaits Expert Wheat Exome |
| サンプル要件 | 高分子量 10 kbp以上、純度 UV260/280 1.7~1.9、DNA量 1000 ng以上 |
| サービスに含まれる内容 | - QC、フラグメンテーション、ライブラリ調製、マルチプレックスでのキャプチャ、AVITIでの1サンプルあたり18 Gbp、PE150のシーケンシング
- 要件を満たすサンプルのライブラリ調製のパフォーマンスコミットメント
- QCレポートより、どのサンプルを残すか、廃棄するか、追加するかを決定することができます
|
▲ リストへ戻る
- Community Capture パッケージ
ターゲットキャプチャキット コミュニティパネル myBaits Custom – Predesigned Community Panelsをご選択の場合*、さらにStandard DNAのサンプル要件を満たす場合ご利用いただけます。
(*Wheat Promoters - Wheat regulatory elements panelは対象外です。)
分解DNAの場合、追加料金でご対応いたします。
コミュニティパネルはこちらよりご確認ください。
| Base DNA Package | 高品質のDNA |
| サンプル要件 | 高分子量 10 kbp以上、純度 UV260/280 1.7~1.9、DNA量 500 ng以上 |
| サービスに含まれる内容 | - QC、フラグメンテーション、ライブラリ調製、マルチプレックスでのキャプチャ、AVITIでの1サンプルあたり1 Gbp、PE150のシーケンシング
- 要件を満たすサンプルのライブラリ調製のパフォーマンスコミットメント
- QCレポートより、どのサンプルを残すか、廃棄するか、追加するかを決定することができます
|
| Standard RNA | 高品質のRNA |
| サンプル要件 | 純度 UV 260:280 1.9~2.1、RNA量 250 ng以上 |
| サービスに含まれる内容 | - QC、DNase処理、精製、directionalライブラリ調製、マルチプレックスでのキャプチャ、AVITIでの1サンプル当たり1Gbp、PE150のシーケンシング
- ヒト・マウス・ラットサンプルでは、mRNAキャプチャとrRNA除去のオプション
- 要件を満たすサンプルのライブラリ調製のパフォーマンスコミットメント
- QCレポートより、どのサンプルを残すか、廃棄するか、追加するかを決定することができます
|
▲ リストへ戻る
ページの先頭に戻る
アプリケーション
- ジェノタイピング、マーカーリシーケンシング、マーカーの発見
- 非モデル生物における変異の発見
- 植物標本館、古代、博物館のDNAシーケンス
- 系統発生的に有益な領域のシーケンス(例: UCE. Mito)
- 耐性遺伝子シーケンシング(RenSeq)
- PacBioおよびOxford Nanoporeのロングインサートターゲットシーケンシング
- メチル化解析用のターゲットエンリッチメント
|
| Marchelら(2018)はMutRenSeqを利用して六倍体コムギ(Triticum aestivum)の黄さび病に対する抵抗性を与える遺伝子を同定しました。Non canonicalなN末端zinc finger BEDドメインを含む、染色体2B上の遺伝子クラスターの3つの遺伝子(Yr7, Yr5, YrSP)を特徴づけました。 |
| 
Marchal, C., Zhang, J., Zhang, P. et al. (2018). BED-domain-containing immune receptors confer diverse resistance spectra to yellow rust. Nature Plants 4: 662–668 |
|
| Ruane & Austin (2017)は、これまでにシーケンスされていなかったXylophis sternohynchus(セタカヘビ科の一種)を含む、ホルマリン保存された10のヘビサンプルのUltraconserved elements (UCEs, 超保存エレメント)を標的としました。現代のヘビを含む系統樹にホルマリン保存したサンプルを配置し、予想される分類学的関係を復元しました。 |
| 
Ruane, S. and Austin, C.C. (2017). Phylogenomics using formalinfixed and 100+ year- old intractable natural history specimens. Molecular Ecology Resources 17: 1003-1008 |
ページの先頭に戻る
FAQ
カテゴリ内開閉 -
なぜDaicel Arbor BiosciencesのNGSサービスは選択されていますか?
 我々myReadsチームは、共同研究者の一人としてプロジェクトに協力しているかのように、NGSサービスを提供することを目標にしています。NGSプロジェクトへ透明で協力的なアプローチを目指すよう、努めています。 我々myReadsチームは、共同研究者の一人としてプロジェクトに協力しているかのように、NGSサービスを提供することを目標にしています。NGSプロジェクトへ透明で協力的なアプローチを目指すよう、努めています。
オープンなコミュニケーションを取り、個々の研究デザインやご要望にあった適切なソリューションを見つけます。 ターゲットキャプチャ、ハイスループットシーケンス、難しいサンプルのプロジェクトなどの分子生物学実験の数十年に渡る集約的な経験があります。 |
どのようなサービスを提供していますか?
 標準的なサンプルから、古代DNAなどの劣化・分解したサンプルまで、次世代シーケンスのフルサービスを提供しています。 サービスには次の内容が含まれます。 標準的なサンプルから、古代DNAなどの劣化・分解したサンプルまで、次世代シーケンスのフルサービスを提供しています。 サービスには次の内容が含まれます。
- Ion Torrent, Illumina, PacBio用のライブラリ調製
- myBaitsの既存パネルおよびカスタムキットを利用したターゲットエンリッチメント
- Ion Torrent Illumina, PacBioの装置を利用したハイスループットシーケンス
|
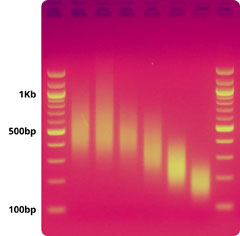
|
| 図. myReads NGSサービスで利用可能なNGSライブラリインサートサイズ選択
Daicel Arbor Biosciencesは、myBaitsターゲットキャプチャキットを利用した特定のプロジェクトに適した最適な平均インサート長を選択するお手伝いをします。特定のプロジェクトには、SNPリシーケンシング、エクソンキャプチャ、UCEシーケンシングなどがあります。
DNAの品質、希望される標的領域の性質、利用するシーケンスプラットフォームやリードプロトコルに応じ、あなたのプロジェクトにおいて、NGSライブラリのサイズを短くしたり、長くしたりすることで、メリットが得られることがあります。 |
サンプルを提出するにあたり、どのような要件がありますか?
 サンプル調製ガイド をご覧ください。 サンプル調製ガイド をご覧ください。
このガイドラインには、標準的なDNAまたはRNA、新鮮かつ高品質の比較的最近のサンプルに由来する抽出物に対する内容が書かれています。
例: 新鮮、または凍結、アルコール保存された組織から抽出されたDNAなど
また、標準的でない、または壊れやすいサンプル、例えば、分解された標的がレアなものなど多くのサービスプロジェクトで受理しています。
例: 古代DNA、博物館、アーカイブ、メタゲノミクス、環境DNAなど
ただし、お問い合わせ頂く最初の段階で、サンプルと研究プロジェクトの詳細をお教えください。プロジェクトを受け入れ可能か、判断致します。その内容に合わせたサンプル提出の要件をお願いすることがあります。 |
ガイドラインで求められる量より少ないDNA/RNAでサービスを実行することはできますか?
 はい、可能です。 はい、可能です。
ただし、Low inputおよび、低品質のサンプルにより、サンプルまたはターゲットにおいて、ドロップアウトのリスクが高くなることにご注意ください。 さまざまなタイプのプロジェクトにサービスを提供してきた経験において、ガイドラインに記載された推奨量が、最良の結果を生んでいます。 |
myReadsプロジェクトの最小のサンプル数は何個ですか?
 最少のサンプル数はありません。 最少のサンプル数はありません。
ただし小規模なプロジェクトは大規模なプロジェクトと比較し、サンプルあたりのコストが高くなることがあります。 大規模プロジェクトでは、サンプルバッチ処理を行い、サンプルあたりの労力が減るため、ボリュームディスカウント価格をご提供しています。 |
納期と保証するカバレッジ深度(coverage depth)についてどのような保証を提供していますか?
 ターゲットキャプチャとハイスループットシーケンスの実験には変動する要素が含まれるため(特にテストを初めて行うような新しいカスタムmyBaits セットなど)、納期や固有のリードのシーケンスカバレッジ深度、On-targetの割合、実験結果などのサービスに関する保証はできません。 ターゲットキャプチャとハイスループットシーケンスの実験には変動する要素が含まれるため(特にテストを初めて行うような新しいカスタムmyBaits セットなど)、納期や固有のリードのシーケンスカバレッジ深度、On-targetの割合、実験結果などのサービスに関する保証はできません。
透明性のある共同プロジェクトを促進するため、全てのカスタムプロジェクトは完全に固有のものであるため、全てのサービスプロジェクトに特定の保証を影響していないことを選択しています。
サイエンスでは、研究プロジェクトが計画通りに100%進まない場合があります。またターゲットキャプチャの場合、事前の実験データなしに特定のシーケンスの結果を予測することは困難です。
ただし、お客様は実行するサービスに対してのみ料金を支払い、いずれのオプションについてもお客様と話し合う前にプロジェクトを受け入れることはありません。 プロジェクトが開始された後は、プロジェクトの進捗について各ステージで更新した情報を御案内します。特定の期限がある場合、それに対応するためにあらゆる努力をします。ただし、その期限までにプロジェクトを完了することを保証するものではありません。 |
サンプルDNAやRNAではなく、ターゲットキャプチャのためのNGSライブラリを提出することはできますか?
 ターゲットキャプチャ用のライブラリの均一性と品質を直接コントロールすることができるため、myReadsチームでのライブラリ調製が好ましいですが、 お客様が作製されたライブラリをご提供いただくこともできます。 ターゲットキャプチャ用のライブラリの均一性と品質を直接コントロールすることができるため、myReadsチームでのライブラリ調製が好ましいですが、 お客様が作製されたライブラリをご提供いただくこともできます。
ライブラリ調製を行われる際はライブラリの種類をお教えください。ターゲットキャプチャの際のアダプタ―特異的な正しいブロッキングオリゴを選択するためと、キャプチャ後の適切な増幅用プライマーを選択するためです。 |
複数の異なるBaits setを利用することはできますか?
 はい、できます。 はい、できます。
ただし、単一のNGSライブラリセットに複数の異なるキットを使用し、キャプチャを行う場合、そのために十分な量が必要になります。 詳細な情報や価格設定については、個別にお問い合わせください。 |
DNA抽出サービスは提供していますか?
 現在、標準サービスのオプションとしてDNA抽出を提供しておりません。 現在、標準サービスのオプションとしてDNA抽出を提供しておりません。
ただ、標準的なサンプル、ならびに分解したサンプルの両方からDNAを抽出するための設備と専門知識を有しています。従い、状況や事情により提供できることがあります。特別なサービスオプションについては直接お問い合わせください。 |
どのような種類のDNAサンプルを受け入れていますか?(ヒト、植物、動物、分解されたDNAなど)
 あらゆる種類の ‘non-hazardous’ DNA抽出物を受け入れています。高品質であるか、分解されたサンプルから抽出したDNAであるかは問いません。 あらゆる種類の ‘non-hazardous’ DNA抽出物を受け入れています。高品質であるか、分解されたサンプルから抽出したDNAであるかは問いません。
ただし、サンプルはサービスプロジェクトの種類に応じた標準の提出要件を満たしている必要があります。該当する場合は、ネガティブコントールを含める必要があります。要件を満たしていない場合は、サンプルが品質管理をパスせず、プロジェクトの遅延に繋がります。
現在ヒトDNAのターゲットキャプチャのためのヒト古代DNAサンプルに関するプロジェクトを受け入れていないことのみ、ご了承ください。 |
ページの先頭に戻る
Publication
ページの先頭に戻る