- 同一のプラットフォーム上で遺伝子、ゲノム、クローン、mRNA、DNA バリアントライブラリ、
Illumina NGS用ライブラリの構築が可能。 - 24時間以内に最大32種類 (BioXp 3250モデル) / 96種類 (BioXp 9600モデル) の高品質dsDNA フラグメントを生成。
- 24時間以内に最大16種類のde novo mRNA※、DNAテンプレートから最大48種類のmRNAを生成。
- 最大7.2kbのクローン化 de novo DNAフラグメント、DNAテンプレートから最大24kbのDNAフラグメント※を合成可能。
- 最大1.8kbのde novo mRNA※、DNAテンプレートから最大10kbのmRNAを合成可能。
- DNAテンプレートから最大37kbのプラスミドの増幅、最大1.8kbのde novo DNA増幅が可能。
- 標準の96ウェルプレートを使用
- 独自の2 段階エラー訂正プロセスによりエラー率 1:10,000 ~1:30,000 bp
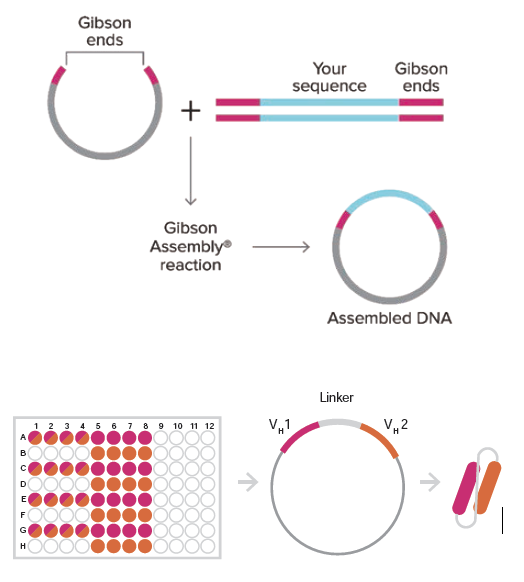
- クローニング効率の高いGibson Assembly®メソッドを使用し、既製またはカスタムのベクターへのクローニングを自動化
- 手動でのプログラム入力は不要。装置内に配置するモジュールからバーコードが自動スキャンされることで、プロジェクト固有のプログラムで自動的に実験が開始されます。
- 等温および温度勾配の両方のアプリケーションに使用可能なサーマルサイクラーを搭載
- 内蔵ソフトウェアによる装置の制御が可能(制御用の別PC は不要)
※現在 BioXp 3250 モデルのみで可能です(2025年3月時点)。詳細は
こちらをご覧ください。
- Introducing the BioXp® 9600 system
など
 | |  | |  |
| 遺伝子の合成 | DNAのクローニング | DNAの増幅 |
| | |
| 下流のプロセスですぐに使用可能な、カスタム遺伝子フラグメントを合成できます。 | 任意のベクターに最大7,200bpのカスタムde novo DNAフラグメントを組み込みクローニングします。テンプレートDNAからは最大24kbのDNAフラグメント合成※が可能です。 | セルフリーでのDNA増幅が可能です。DNAテンプレートからは最大37kbのプラスミド増幅が可能です。 |
| | | | |
 | |  | |  |
| mRNAの合成 | ライブラリ構築 | NGSライブラリ調製 |
| | |
| de novo mRNA 最大1,800bp※まで、テンプレートDNAから最大10kbまでのmRNAを合成できます。 | カスタムバリアントDNA ライブラリ(スキャニングライブラリ、コンビナト リアルライブラリ)の合成が可能です。 | Illumina NGS用のライブラリ調製が可能です。 |
※現在はBioXp 3250モデルのみで可能です。
| モデル | BioXp® 3250 | BioXp® 9600 |
アプリケーション
プロジェクト | スルー
プット | サイズ
(kb) | ラン
タイム
(時間) | スルー
プット | サイズ
(kb) | ラン
タイム
(時間) |
| Gene synthesis | 最大32 | 0.3 - 1.8 | ~14-16 | 最大96 | 0.3 - 1.8 | ~14-16 |
| De novo DNA Cloning | 最大32 | インサートDNA:
0.3‒7.2
ベクターDNA:
2.0-12 | ~12-21 | 最大96 | インサートDNA:
0.3‒7.2
ベクターDNA:
2.0-12 | ~12-21 |
Select DNA Cloning Kits,
Gibson Assembly®
(DNAテンプレート使用) | 最大24 | 0.3 - 24
(インサートDNA:
0.3-6.0 kb x4
fragsまで) | 2 | |
Select DNA Cloning,
Golden Gate Assembly
(DNAテンプレート使用) | 最大24 | 0.15 - 9.0
(インサートDNA:
0.15-1.8 kb x5
frags まで) | ~2
(1-4 frags)
~6.5
(5 frags) | |
| De novo DNA cloning and amplification | 最大22 | インサートDNA:
0.3-1.8
ベクターDNA:
2.7-12 | ~24.5 | 最大22 | インサートDNA:
0.3-1.8
ベクターDNA:
2.7-12 | ~24.5 |
Select plasmid
amplification
(DNAテンプレート使用) | 最大24 | 1.8 - 37 | 7 | 最大96 | 1.8 - 37 | 7 |
Select DNA cloning and amplification
(DNAテンプレート使用) | 最大24 | インサートDNA:
0.3-1.8
ベクターDNA:
2.7-12 | 10 | 最大96 | インサートDNA:
0.3-1.8
ベクターDNA:
2.7-12 | ~11 |
De novo mRNA
synthesis | 最大16 | 0.4 - 1.8 | ~18 | |
Select mRNA synthesis
(DNAテンプレート使用) | 最大48 | 0.4 - 10 | ~4-6 | 最大48 | 0.4 - 10 | ~4-6 |
| DNA Libraries | 最大32 | 0.3 - 0.8 | ~16.5 | 最大96 | 0.3 - 0.8 | ~16.5 |
| Whole Genome Sequencing library prep | 最大24 | インプットDNA:
10-20 ng | | 最大96 | インプットDNA:
10-20 ng | |
| NGS library prep for plasmid sequencing | 最大24 | インプットDNA:
10-20 ng | | 最大96 | インプットDNA:
10-20 ng | |
アプリケーションプロジェクトの詳細は
こちらをご覧ください。
 プレゼンテーション@mRNA-Based Therapeutics Summit EUROPE
プレゼンテーション@mRNA-Based Therapeutics Summit EUROPE 
『Engineering and Results of Next-Generation COVID-19 Vaccines』
次世代COVID-19ワクチンのエンジニアリングと結果
-自動化の力を活用して、ワクチンの迅速な合成と発見を実現する-
| |
Codex DNAが主催するこの講演では、RNAimmuneのR&DアソシエイトディレクターであるDavid Brown博士が、mRNAワクチン開発プログラムを推進する主要なテクノロジー、ツール、およびノウハウを紹介します。彼は、チームがBioXp™システムを利用して、スクリーニング用のワクチン候補を迅速に生成し、コンセプトから発見までの時間を短縮するのに役立つ自動化ワークフローを開発した方法について説明します。
この短い講演(約15分)では、ブラウン博士が次のトピックに触れます。
- COVID-19ワクチンの設計と合成
- 前臨床データからのいくつかのSARS-CoV-2株にわたる広範な中和
- 自動化されたBioXp™システムを使用したCOVID-19ワクチン変異体の迅速な合成およびクローニングワークフロー
BioXp
®には、様々なアプリケーションプロジェクトが用意されています。
選択したアプリケーションプロジェクトに応じた消耗品キット(モジュール)が提供されます。
 BioXp® gene synthesis kit BioXp® gene synthesis kit |
| 下流のプロセスですぐに使えるdsDNAを合成します。 |
仕様:
- 最大32rxn/run (BioXp 3250)
最大96rxn/run (BioXp 9600) - 収量: >200ng/rxn
- サイズ:0.3 ‒ 1.8※kb
- 20 ~70% のGC 含量
- 最小注文:1 遺伝子
- システムランタイム: ~14-16 時間
- Codex DNA ベクターまたは独自のベクターに適応
- pUCGA ends またはカスタムends が選択可能
※1.8kb以上3.0kb未満もご注文は可能ですが、製造できないことがあります。
|  |
| ※使用モジュール:A・B |
 BioXp® De novo DNA cloning kit BioXp® De novo DNA cloning kit |
1~4つのde novo DNAフラグメントを選択したベクターにシームレスに組み立て、すぐに形質転換に使えるDNAをクローニングします。
|
仕様:
- 最大32rxn/run (BioXp 3250)
最大96rxn/run (BioXp 9600) - インサートサイズ:0.3-7.2 kb
(最大1.8 kb のde novo フラグメント4 つ+ リンカー領域を結合可能)
ベクターサイズ:2.0-12 kb - 収量:>200 ng/rxn
- 1ランで最大4種類のベクターを用いたクローニングが可能
- システムランタイム:~12-21時間
(新規のDNA 合成からクローニングまで) - 残った未使用のフラグメント製品を使用可能
|  |
| ※使用モジュール:A・B・C |
| ▲ アプリケーションプロジェクトの先頭へ戻る |
 BioXp® Select DNA Cloning kit, Gibson Assembly® BioXp® Select DNA Cloning kit, Gibson Assembly® |
ユーザー提供のベクターとDNAフラグメントから、1度の操作で自動的にDNAクローニングが可能なキットです。※BioXp 3250モデルのみ対応
|
仕様:
- アウトプット: 環状二本鎖DNAクローン
- インプット:以下のDNAフラグメント(1~4フラグメント)とベクター
・BioXp Gene Fragments (300-1800bp)
・BioXp Variant Libraries (300-800bp)
・IDT gBlocks™ (300-3000bp)
・Twist Genes (300-1800bp)
・PCR アンプリコン (300-6000bp)
・ベクター:Telesis Bio社提供のベクターまたはユーザー提供のベクター
※ベクターとインサートには40bpの相同配列、各インサートは80bpの相同配列が必要です。 - スループット: 8, 24rxn/run
- システムランタイム: 2時間
|
| ※使用モジュール:A・C・J |
| ▲ アプリケーションプロジェクトの先頭へ戻る |
 BioXp® Select DNA Cloning kit, Golden Gate Assembly BioXp® Select DNA Cloning kit, Golden Gate Assembly |
最も一般的な2種類のIIS制限酵素であるBsaIおよびBsmbIを使用し、ユーザー提供のベクターとDNAフラグメントから、1度の操作で自動的にDNAクローニングが可能なキットです。※BioXp 3250モデルのみ対応
|
仕様:
- アウトプット: 環状二本鎖DNAクローン
- インプット:以下のDNAフラグメント(1~5フラグメントを推奨)とベクター
・BioXp Gene Fragments (300-1800bp)
・IDT gBlocks™ (300-1800bp)
・Twist Genes (300-1800bp)
・PCR 産物 (150-1800bp)
・あらかじめクローン化されたインサートを含むエントリーベクターもサポート。
・ベクター:Telesis Bio社提供のベクターまたはユーザー提供のベクター - スループット: 8, 24rxn/run
- システムランタイム: ~2 時間 (1-4 frags)、~6.5 時間 (5 frags)
|
| ※使用モジュール:A・J・L |
| ▲ アプリケーションプロジェクトの先頭へ戻る |
 BioXp® De novo DNA cloning and amplification kit BioXp® De novo DNA cloning and amplification kit |
DNAフラグメントをベクターに組み込んだ後に、dsDNAフラグメントの増幅を行います。形質転換にすぐに使えるDNAを1日で合成し、2日目には哺乳類細胞のトランスフェクションを可能にすることで、遺伝子発現研究を促進します。
|
仕様:
- 収量:>10µg DNA/rxn
- サイズ:インサートDNA:0.3-1.8kb、ベクターDNA:2.7-12kb
- 大腸菌形質転換が不要: エンドトキシン汚染を回避、プラスミド準備の時間と費用を排除します。
- システムランタイム:~24.5時間
- スループット:14、22rxn/run
|
| ※使用モジュール:A・C・D・M |
| ▲ アプリケーションプロジェクトの先頭へ戻る |
 BioXp® Select plasmid amplification kit BioXp® Select plasmid amplification kit |
配列検証済みプラスミドからDNA増幅を行うために必要な試薬が含まれています。
|
仕様:
- 収量:>10µg DNA/rxn
- インプット:プラスミド (サイズ1.8-37 kb)
- アウトプット:増幅DNA (分岐したコンカテマー)
- システムランタイム:7時間 (BioXp 3250、9600)
- スループット:8、16、24 rxn/run (BioXp 3250)
8、16、24、48、96rxn/run (BioXp 9600) - ※使用モジュール:A・I・J
|
| ▲ アプリケーションプロジェクトの先頭へ戻る |
 BioXp® Select DNA cloning and amplification kit BioXp® Select DNA cloning and amplification kit |
フラグメントDNAとベクターからDNA増幅を行うために必要なGibson Assembly®とその他の試薬が含まれています。
|
仕様:
- 収量:>10µg DNA/rxn
- インプット:インサート (直鎖状DNAフラグメント 0.3-1.8 kb)、ベクター (直鎖状 2.7-12 kb)
- アウトプット:増幅DNA (分岐したコンカテマー)
- システムランタイム:10時間 (BioXp 3250)、11時間 (BioXp 9600)
- スループット:8、16、24 rxn/run (BioXp 3250)
8、16、24、48、96rxn/run (BioXp 9600) - 使用モジュール:A、C、I、J(BioXp 3250)/J2(BioXp 9600)
|
| |
| ▲ アプリケーションプロジェクトの先頭へ戻る |
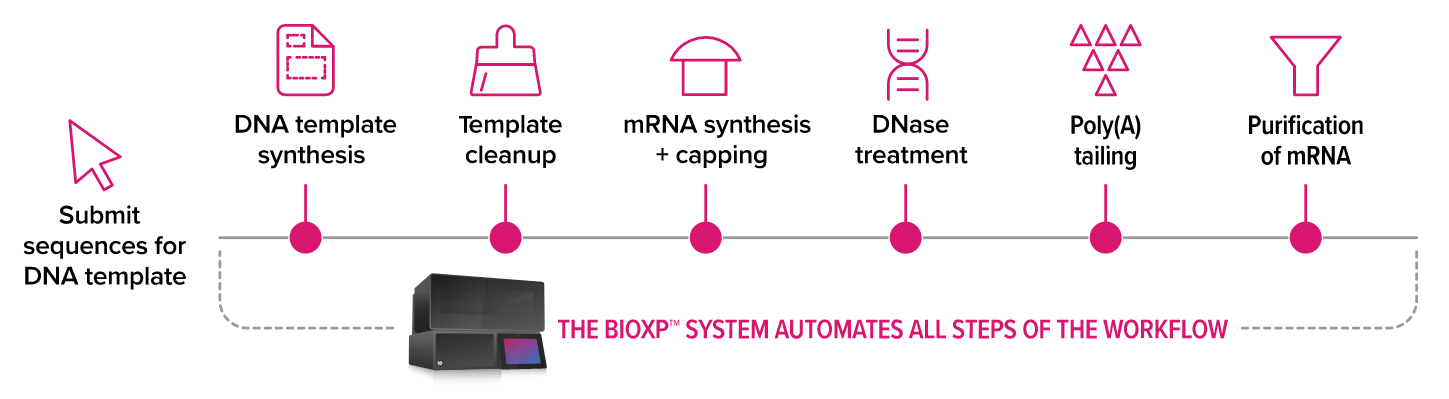
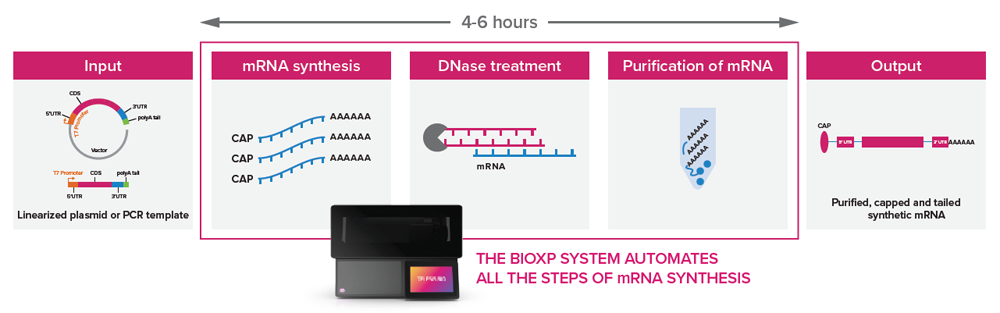
 BioXp® De novo mRNA synthesis kit BioXp® De novo mRNA synthesis kit |
最大 1.8 kb 長のエラー訂正済み遺伝子断片 (DNA テンプレート) と、キャップおよびテール付き合成 mRNA を生成するために必要な試薬がすべて含まれています。オプションとして、mRNAの合成時の修飾ヌクレオシドの有無を選択することができます。※BioXp 3250モデルのみ対応
|
仕様:
- 収量:最低 5μg・中央値 10μg (配列による)
- サイズ:0.4~1.8kb
- 5' CleanCap® AG (3'OMe)キャップ、poly(A)テール(中央値:150As) 付加
- 修飾 (オプション):N1-methyl-pseudouridine
- システムランタイム:~18時間
- スループット:8または16サンプル/run
- 使用モジュール:A、F、G
|
| |
 |
| ▲ アプリケーションプロジェクトの先頭へ戻る |
 BioXp® Select mRNA synthesis kit BioXp® Select mRNA synthesis kit |
直鎖状プラスミドDNA または PCR産物のDNAから、最大 10 kb のキャップおよびテール付き精製 mRNA を合成することができます。合成したmRNAは、トランスフェクションに使用可能です。
|
仕様:
- 収量:>50µg
- サイズ:0.4~10kb
- 5' CleanCap AG (3'OMe)、poly(A)テール付加
- 修飾 (オプション):N1-methyl-pseudouridine
- システムランタイム:~4~6時間 (BioXp 3250、9600)
- スループット:8、24、48 rxn/run (BioXp 3250、9600)
- 使用モジュール:A、F、H
|
| |
 |
| ▲ アプリケーションプロジェクトの先頭へ戻る |
 BioXp® error-collected variant libraries BioXp® error-collected variant libraries |
| |
 | ウェルごとにアミノ酸/コドンを1つずつ置換したシンプル・スキャニング・ライブラリと、縮重コドン(NNN、NNK、NNS)を使用した部位飽和ライブラリを合成可能です。活性を持つアミノ酸位置の特定に有用です。
仕様:
- フォーマット:線形二本鎖 DNA
- サイズ:300 - 800 bp*
- GC含量:30 - 60%**
- 縮重コドン:NNN, NNK, NNS
- 最大縮重コドン数:1配列あたり3(隣接)または6(非隣接)
- スループット:最大32rxn/run (BioXp 3250)
最大96rxn/run (BioXp 9600) - 収量: >200ng/rxn
- 最小注文:32 バリアント
- システムランタイム:~16.5時間
* 長さ2,000bp未満は注文可能ですが、保証できません。
** GC含量20% < X < 70%は注文可能ですが、保証できません。
|
 |
縮重塩基を使用してタンパク質の結合と機能を最適化する、多様な複数の非隣接アミノ酸位置を置換された多種多様なコンビナトリアル・ライブラリを合成可能です。目的の効果に最適なアミノ酸位置の特定に有用です。
仕様:
- フォーマット:線形二本鎖 DNA
- サイズ:300 - 800 bp*
- GC含量:30 - 60%**
- 縮重コドン:NNN, NNK, NNS + IUPACヌクレオチド
- 最大縮重コドン数:1配列あたり3(隣接)または6(非隣接)
- スループット:最大32rxn/run (BioXp 3250)
最大96rxn/run (BioXp 9600) - 収量: >200ng/rxn
- システムランタイム:~16.5 時間
* 長さ2,000bp未満は注文可能ですが、保証できません。
** GC含量20% < X < 70%は注文可能ですが、保証できません。
|
※ライブラリ合成サービスもございます。詳細はお問合せください。
|
※使用モジュール:A・B
|
| ▲ アプリケーションプロジェクトの先頭へ戻る |
 BioXp® NGS Library Prep kit for Whole Genome Sequencing BioXp® NGS Library Prep kit for Whole Genome Sequencing |
Illumina NGS用のライブラリ調製キット。全ゲノムシーケンシング用。
|
仕様:
- アウトプット:Illumina シーケンサー用のアダプター付きNGSライブラリ
- インプット:精製ゲノムDNA(10 - 20 ng)
- スループット: 8、24 rxn /run (BioXp 3250)
8、24、48、96 rxn /run (BioXp 9600) |
| ▲ アプリケーションプロジェクトの先頭へ戻る |
 BioXp® NGS Library Prep kit for Plasmid Sequencing BioXp® NGS Library Prep kit for Plasmid Sequencing |
Illumina NGS用のライブラリ調製キット。プラスミドシーケンシング用。
|
仕様:
- アウトプット:Illumina シーケンサー用のアダプター付きNGSライブラリ
- インプット:精製プラスミドDNA (10 - 20 ng)
- スループット: 8、24 rxn /run (BioXp 3250)
8、24、48、96 rxn /run (BioXp 9600) |
| ▲ アプリケーションプロジェクトの先頭へ戻る |
 BioXp™システムによるSARS-CoV-2のゲノム全体の構築 BioXp™システムによるSARS-CoV-2のゲノム全体の構築 |
SARS-CoV-2は、⻑さ約30 kbの⼀本鎖ポジティブセンスRNAゲノムを持ち、これをRNAウイルスの中で最⼤のものの1つにしています。合成⽣物学、特にDNAライティング技術により、予防薬 (ワクチン) と治療薬 (モノクローナル抗体) の迅速な開発が可能になります。さらに、DNAライティングにより、診断キットおよびアッセイ開発のための品質保証された材料の⽣成が可能になります。合理的に再設計された、完全⻑の弱毒化ゲノムは、このウイルスに対するワクチンの開発を⽀援します。さらに、ウイルスのゲノムパーツはウイルス抗原に対する抗体を増やすのを容易にし、感染した患者を効果的に治療することができる治療的なオプションの開発を可能にします。
BioXp™システムを使⽤してSARS-CoV-2のゲノムパーツを設計し、合成しました。 また、SARS-CoV-2の全⻑ゲノムを⼤腸菌の細菌⼈⼯染⾊体 (BAC) として組み⽴てることもできました。
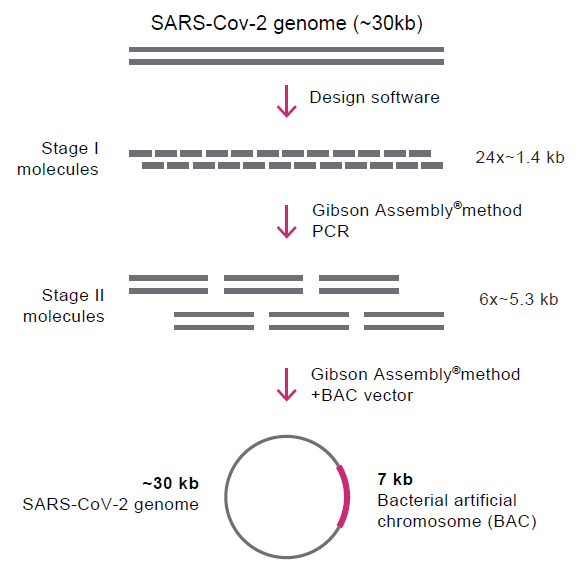 | | 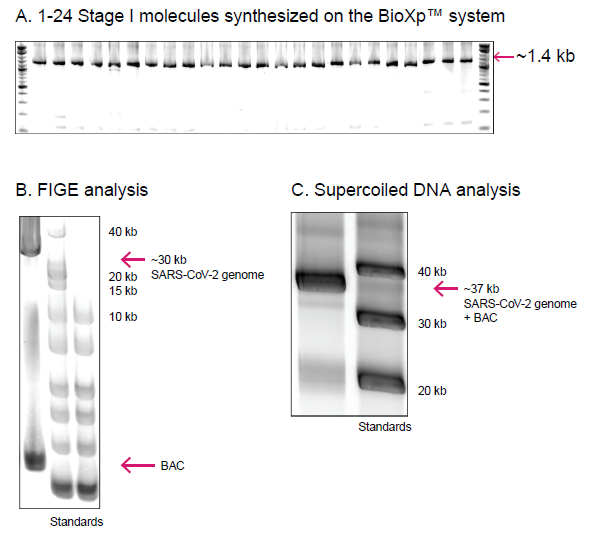 |
| Figure 1. SARS-CoV-2ゲノムの階層アセンブリ・スキーム。 ゲノムは3段階のアセンブリを使⽤して合成された。24X〜1.4 kbのStage I アセンブリがBioXp™システムで完了した後、Stage II 分⼦への6X〜5.3 kbアセンブリおよび〜30 kbのStage III ゲノムへのアセンブリが続いた。 | | Figure 2. BioXp™システムを使⽤したSARS-CoV-2ゲノムのアセンブリ。(A) BioXp™システムで合成された24 X Stage I 分⼦を、Stage II および Stage III 分⼦へと組み⽴てた。(B) 制限エンドヌクレアーゼ処理を使⽤してBACから約30 kbのSARSCoV-2ゲノムを切り離した後の、Stage III 分⼦を持つクローンのFIGE解析を⽤いた解析。(C) 分⼦量スタンダードと並べた、BACへとクローン化した無傷なゲノムのSupercoiled DNA解析(単⼀の、代表的なクローンからのDNA解析を⽰す)。 |
Telesis Bio社 Application Note “Harnessing the power of the BioXp™ system to combat COVID-19 caused by SARS-CoV-2” より引用。
※
アプリケーションノート日本語版はこちらからダウンロード可能です。 (簡単な個人情報の登録が必要です)
▲ アプリケーション事例一覧へ戻る
 抗体産⽣の⾃動化・効率化によるターゲット検証速度の向上 抗体産⽣の⾃動化・効率化によるターゲット検証速度の向上 |
BioXp™システムは、遺伝⼦改変T細胞受容体 (TCR) に必要な抗体コンストラクトの構築を合理化・迅速化するとともに、より効率的なターゲットスクリーニングを可能にします。BioXp™システムを⽤いて正確な完全⻑のコンストラクトを構築することで、検証までのサイクルを短縮し、リード候補の特定に関わるプロセスを加速することができます。
あるバイオ製薬企業 (以下、この企業) は、BioXp™システムを⾃社のワークフローに組み込むことで、「Design-Build-Test」のサイクルに要する時間を⼤幅に削減し、ワークフローを6週間から3週間に短縮すると同時に、パイプラインを25%増加させました。
この企業が採⽤していた従来のscFv組み⽴てワークフローは、5〜7⽇かかるものでした。ワークフローの多くのステップが⼿作業に頼っており、どの段階でも不正確なピペッティングやサンプルの取り違えなどのヒューマンエラーが発⽣する可能性がありました。
BioXp™システムのワークフローでは、完全⻑のscFvコンストラクトを⾃動化装置での1回のランで構築することができるため、サンプル数を半減し、コンストラクトの設計を簡素化します。開始から終了まで4〜5⽇しかかからないため、貴重な時間とリソースを節約することができます。コンストラクトの設計に続いて、BioXp™システムで完全⻑のフラグメントを構築し、ベクターとライゲーションします。候補を確認するために、形質転換、コロニーの採取、およびシーケンシングを⾏います。
今回の研究では、作成した93種類のコンストラクトから各2個のコロニーをシーケンシングし、70%以上がエラーのないクローン (EFC) であることが判明しました。このEFCを⽤いて、トランスフェクション、抗体産⽣、スクリーニングを⾏いました。BioXp™システムは、検証までのサイクルを短縮し、ワークフローから多くの⼿動プロセスを除外し、リード候補の同定に関わるプロセスを加速しました。
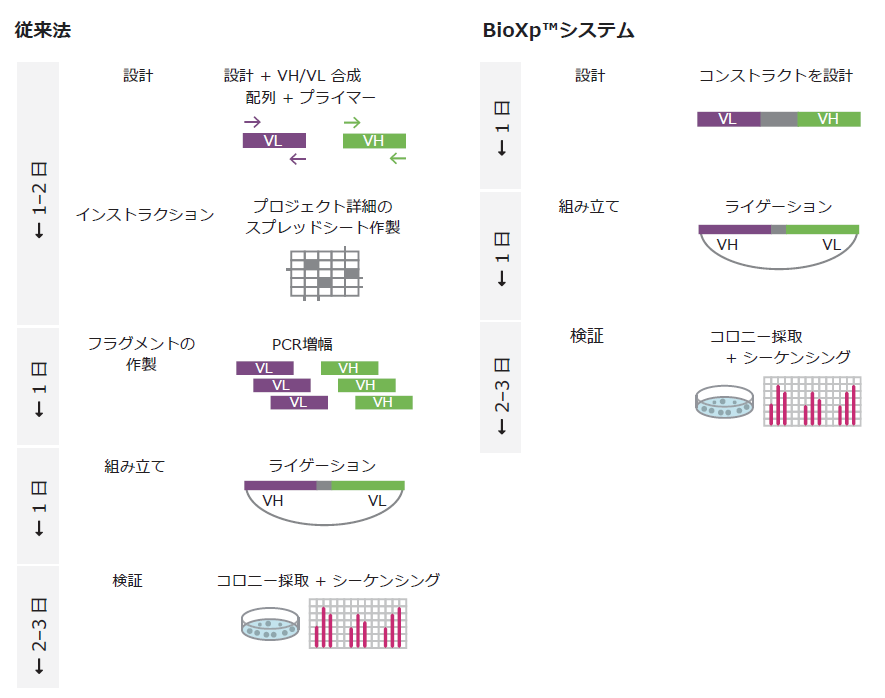
Figure:scFvの組み⽴て。従来法 vs BioXp™システムのワークフロー。
BioXp™システムの⽅法では、クローニングのステップ数が減り、ワークフローを最適化している。
Telesis Bio社 Case Study “Improvements in target validation rates by automating and streamlining antibody production” より引用。
※
アプリケーションノート日本語版はこちらからダウンロード可能です。 (簡単な個人情報の登録が必要です)
▲ アプリケーション事例一覧へ戻る
 BioXp™ 3250システムで構築されたライブラリの検証 BioXp™ 3250システムで構築されたライブラリの検証 |
BioXp™システムでは独⾃の⽅法でdsDNAライブラリを構築するため、下流スクリーニングプロセスをより迅速に開始することができ、「Design-Build-Test」のサイクルに要する時間を⼤幅に短縮することができます。BioXp™システムで構築されたDNAライブラリの品質を実証するために、A社がBioXp™システムで構築した2つのライブラリ・プロジェクトのブラインドデータを紹介します。
NNK スキャニング・ライブラリ構築
タンパク質の機能研究にはスキャニング・ライブラリが欠かせません。A社はまず、BioXp™システムでスキャニング・ライブラリを構築し、構築されたBioXp™ライブラリを回収し、各ウェルの産物を次世代シーケンス (NGS) で解析しました (528,627 reads)。
このライブラリでは、20カ所のアミノ酸位置に対してNNK置換を行いました。NNK置換には32通りのコドンの組み合わせがあるので、20か所の置換では、合計640通りのコドンの組み合わせが得られる可能性があります。
ヒートマップ(Fig.1)とコドン分布 (Fig.2) に⽰すように、BioXp™スキャニング・ライブラリは合成される可能性がある640個のコドンを100%含んでおり、20か所すべてに可能性のあるトリヌクレオチドが存在していることが分かりました。また予想通り、G/Cバイアスも僅かに⾒られました。ヒートマップとコドン分布のグラフから、BioXp™システムが想定されるコンストラクトを全て含んだ高い多様性を持つ20か所のバリアントライブラリを構築したことが確認できました。 | | 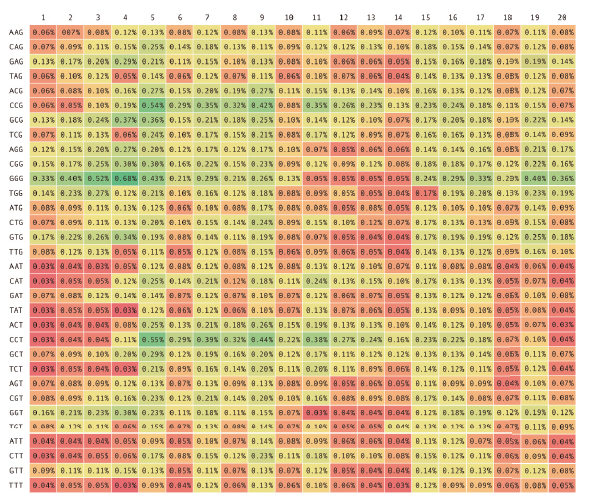
Figure 1. 20か所のNNKスキャンから得られた、640個のコドンの出現率を⽰すヒートマップ。
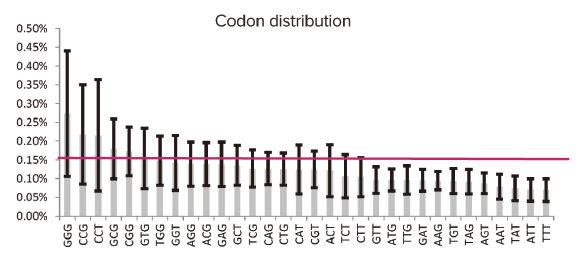
Figure 2. コドン分布。理論上のコドン分布は0.156%。観察されたコドン分布は、G/Cに偏っている。 |
コンビナトリアル・ライブラリ構築28 bpの長さのDNAについて、タンパク質中の以前に同定された機能性領域に関連することが知られている塩基10カ所を、IUPAC letters を用いてランダムな塩基に置換しました。発現した変異タンパク質の様々な物理的特性や酵素活性を評価しました。
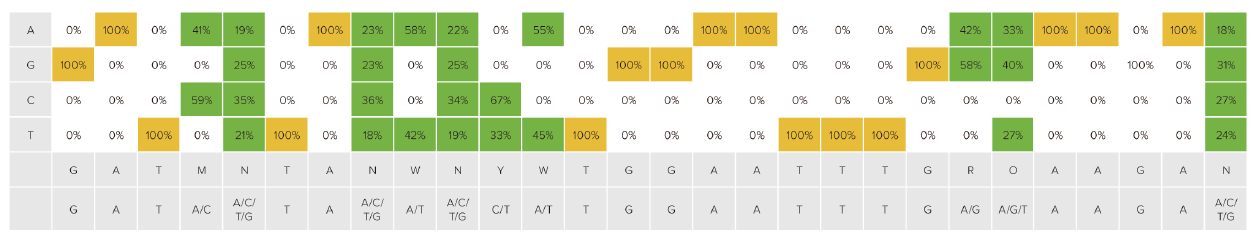
Figure 3.IUPACコードを⽤いて、28bpの遺伝⼦中の10個の塩基を変化させたバリアントライブラリ。
※⻩⾊は変異を⼊れていない部分。緑⾊はIUPAC Lettersに従ってランダムに塩基を置換した位置。NGSの結果、実際に置換された塩基の種類を%で⽰している。
Fig.3は、今回のライブラリ構築で変異させた10カ所の位置の配列分布を⽰しています。すべての⾮変異配列を⻩⾊で⽰しており、NGSの結果、BioXp™ライブラリのこれらの⾮変異位置の配列が完全 (元配列の塩基と合致)であることが確認されました。変異を⼊れた位置は緑⾊で⽰しています。NGSの結果、Fig.3に⽰すように、各変異位置に各変異塩基がほぼ同じ割合で存在していることが⽰されました。このことから、BioXp™ライブラリが⾼い多様性を持つことが分かります。
典型的なライブラリ構築と同様に、8、10、11番⽬のシトシンの取り込みにわずかな偏りがありましたが、全体的に⾒て、BioXp™ライブラリは全ての変異位置において、ほぼ予想通りの⽐率で予想通りの変異を含んでいることが分かりました。また、NGS 586,343 readsのうち、549,230 (94%) がユニークでした。
Telesis Bio社アプリケーション資料 ”BioXp™ variant libraries” より引用。
※
アプリケーションノート日本語版はこちらからダウンロード可能です。 (簡単な個人情報の登録が必要です)
▲ アプリケーション事例一覧へ戻る
 ワクチン開発のための効率的なナノボディ・バリアントライブラリの構築 ワクチン開発のための効率的なナノボディ・バリアントライブラリの構築 |
ラクダ科やサメ科の動物は、従来の軽鎖を持たない重鎖抗体 (HCAB) と呼ばれるユニークなクラスの抗体を持っています。これらのシングルドメインの抗原結合部位 (VHHまたはナノボディと呼ばれる) はサイズが⼩さい (12〜14kDa) ので、バイオ医薬品的な観点からその有⽤性への関⼼が徐々に⾼まっています。
Depickerの研究室ではBioXp™システムを⽤いる以前、⼿作業でクローニングを⾏い抗体のバリアントを作製していましたが、BioXp™システムを採⽤することで、バリアント構築を効率化しました。
⼿動で⾏うワークフローには2ステップを要し、まず30種類のバリアントフラグメント(VHH) を合成してベクター1にクローニングし、さらにFc領域を合成してベクター2にクローニングします。次に、それぞれのコンストラクトについて細菌の形質転換を⾏い、その後、プレーティング、コロニーの採取、培養、プラスミドの調製、配列の検証を⾏います。最終的な発現コンストラクト (VHHフラグメントとFc領域を融合させたVHH-MGコンストラクト) は、2ラウンド⽬のクローニングで組み⽴てられます。第2ラウンド⽬のクローニングにおいて、1ラウンド⽬のクローニングで⾏った全てのステップ (最終的な配列決定ステップを含む) を繰り返すことで、タンパク質の発現とバリアント解析のためのP. pastorisへの形質転換が可能となります。
BioXp™システムの場合、まず、バリアント配列をCodex社のDNA注⽂ポータルに登録することで、カスタマイズされた試薬が調製されてエンドユーザーに納品されます。この試薬はGibson Assembly®による⾃動化クローニング専⽤に設計されており、最終的なコンストラクトをシームレスに組み⽴てるために必要な、インサートバリアントとベクター間の40bpの相同オーバーラップが含まれています。さらに、BioXp™システムのワークフローでは、2段階のエラー補正と精製ステップによってエラーの発⽣を最⼩限に抑えます。このように、BioXp™システムでは、⼿作業のクローニングと⽐べてより早く、効率的に、かつ⾼い忠実度でバリアントを作製することできました。
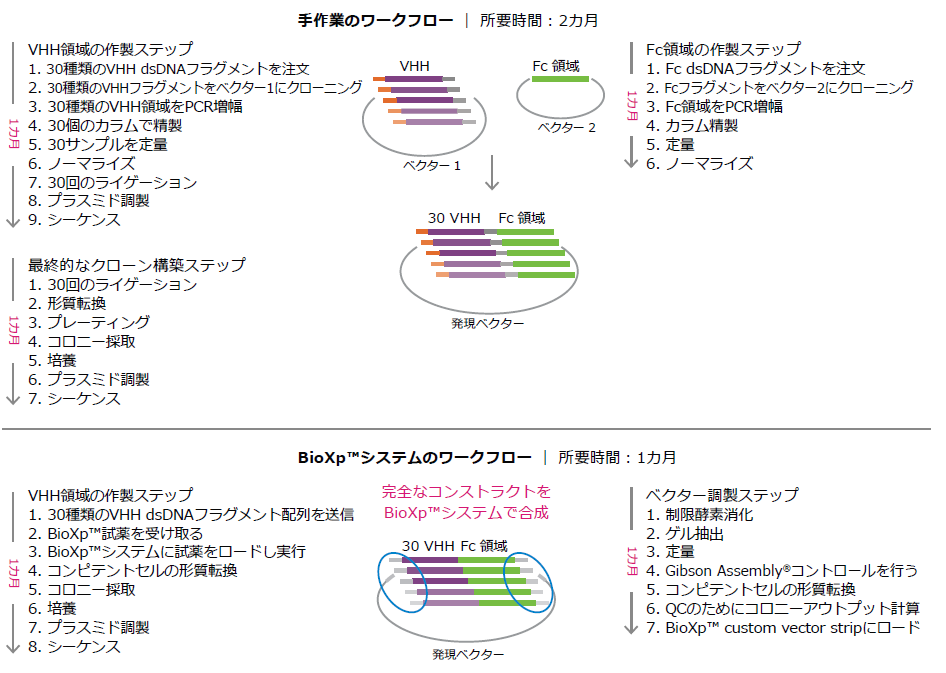
Figure: VHHバリアントの作製に必要な⼿作業のワークフローとBioXp™システムのプロトコルの⽐較
Telesis Bio社 Case Study “Obtain variant constructs one month faster than using manual cloning” より引用
※
アプリケーションノート日本語版はこちらからダウンロード可能です。 (簡単な個人情報の登録が必要です)
▲ アプリケーション事例一覧へ戻る
全自動合成生物学ワークステーション「BioXp™3250」のご紹介
~抗体医療などのバイオ医薬品開発のボトルネック解消に貢献~ (ライブ実施日時:2023/5/23)


合成生物学(Synthetic Biology)とは、in silico上で生物のゲノムを人工的にデザインすることを目的としたコンピュータ工学と遺伝子工学を融合させた学問分野です。近年では抗体医薬品開発や特にウイルスワクチン開発などの領域において注目されています。
BioXp
™3250はGibson Assembly技術を用いることによってDNA断片合成をはじめとした各種遺伝子合成をオートマチックで行い、合成生物学を用いたバイオ医薬品開発の発展を加速させます。
ベクターの情報を外部に出さずにインサート配列をシームレスかつハイスループットにクローニングできるだけではなく、縮重塩基を用いた変異体ライブラリの構築やデジタル配列をもとにした全自動mRNA合成などのバリエーションに富んだアプリケーションを専用ポータルサイトから簡単に設計・注文が可能です。
本ウェビナーでは、BioXp
™3250が提供する合成生物学における全自動ワークステーションとしての特徴と応用例について、抗体・医薬品開発に関する実用例を交えてご紹介します。
※上記画像をクリックいただくと、別タブで視聴ページが開きます。お名前等をご入力の上視聴ください。
抗体医薬品開発・免疫療法のための遺伝子合成ワークステーション
~クローニングからmRNAの全自動合成まで~ (ライブ実施日時:2021/8/27)

BioXp™3250は、DNAやmRNAといった遺伝子の全自動合成を可能にし、合成生物学における全自動ワークステーションとしての統合されたプラットフォームを提供します。ご自身のベクター情報を外部に出すことなく300~7,200 bpのインサート配列をハイスループットにクローニング出来る他、1.8kbのmRNAの全自動合成、多様なライブラリプロジェクトなど、統合されたポータルサイト上から簡単に設計・発注が可能です。
本セミナーでは、BioXp™3250の特長と応用例について、抗体開発研究などのケーススタディを交えながらご紹介させていただきます。
※上記画像をクリックいただくと、別タブで視聴ページが開きます。お名前等をご入力の上視聴ください。
Harnessing the power of the BioXp™ system to combat COVID-19
上記画像をクリックすると、詳細ページが開きます。

[Click here to access]ボタンより、お客様情報をご入力いただくと視聴画面が開きます。(動画の長さ:約38分)
このウェビナーでは、Codex DNAの科学者が、COVID-19ワクチン、治療法、および診断薬の開発をサポートするため、前例のない速度でSARS-CoV-2 の完全長ゲノム変異体をクローン化するために、世界で唯一の完全自動化されたベンチトップ合成生物学ワークステーション:BioXp
™システムと、業界標準のGibsonAssembly
®メソッドをどのように活用したかを共有しています。
※ライブ実施:September 21, 2021 | 11:00 am - 12:00 pm EDT