MERFISH技術は、ハーバード大学 Xiaowei Zhuang 博士の研究室で開発された、空間分解シングルセル・トランスクリプトームプロファイリング技術です *。単一分子FISH (smFISH) の機能を拡張し、ラベリングの組み合わせ、連続的なイメージング、エラーロバスト (エラーに対する頑健性が高い) なバーコーディングを用いて、ミクロン以下の解像度でRNAを検出します。
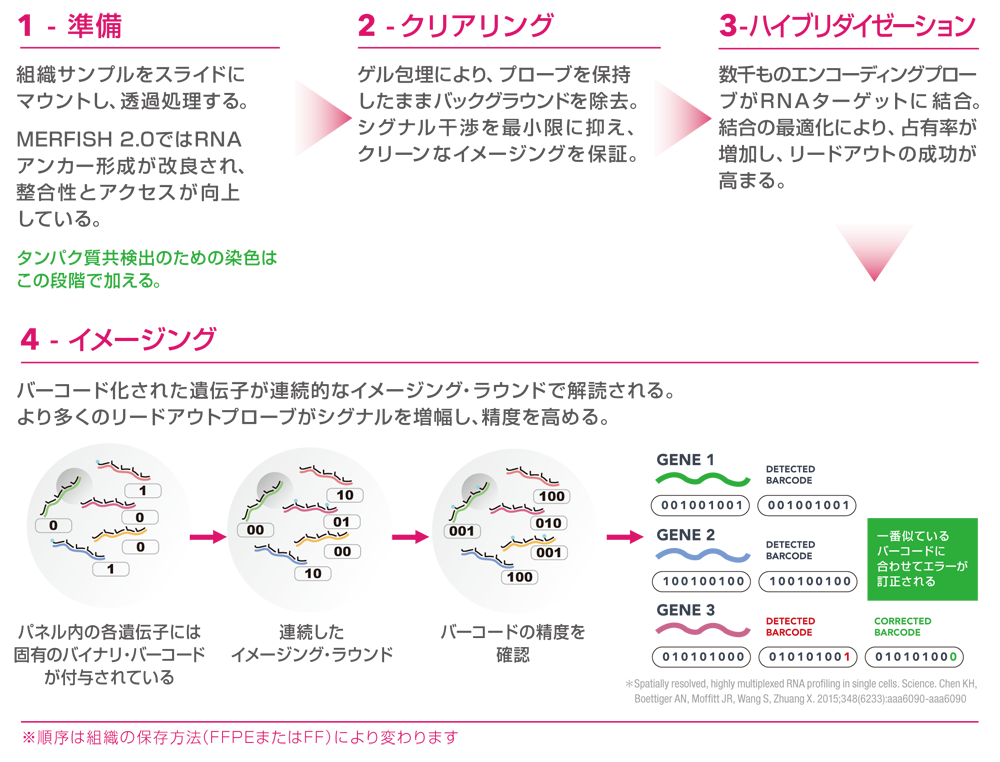
MERFISH測定では、各遺伝子に固有のバイナリ・バーコードを割り当て、smFISHの連続したラウンドを使用してバーコードを光学的に検出します。各イメージングラウンドにおいて、対応するビット位置に1を含むバーコードを割り当てられた遺伝子はsmFISHとして現れ、0ビットを含むバーコードを割り当てられた遺伝子は暗いままとなります。このsmFISHスポットのパターンにより、数百から数千の転写産物を空間的に分解することができます。
万が一、バックグラウンドの蛍光が読み取りエラーを起こした場合でも、システムは簡単に最も近い正しいバーコードに読み取り値を割り当てることができます。このシステムのエラー訂正型バーコーディングとラベリングの組み合わせにより、転写産物の最高の検出精度が保証されます。
これにより、研究者はすべての細胞内のトランスクリプトームの細胞内局在を見ることができるようになります。
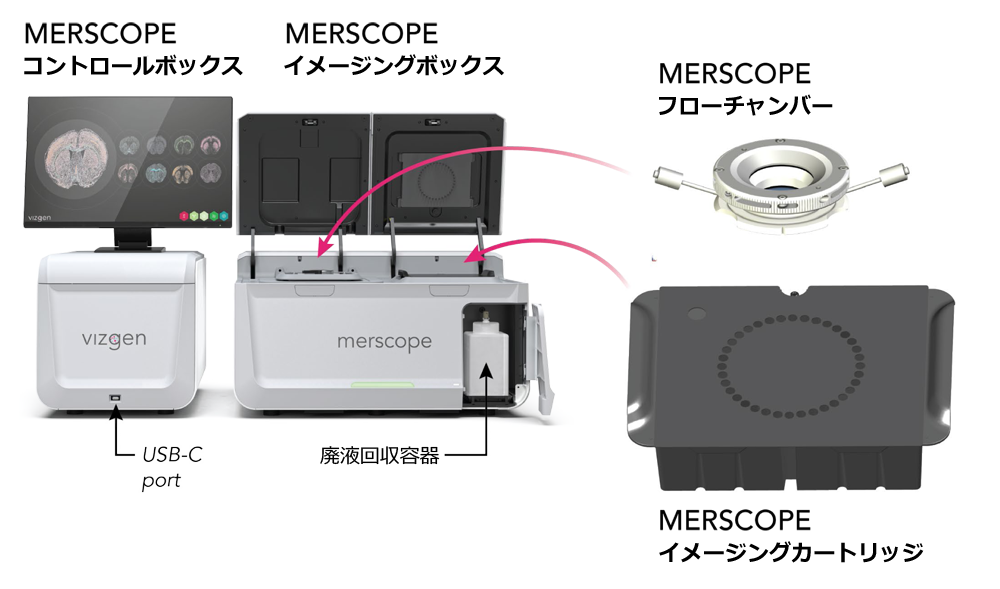
取得されたデータは、MERSCOPE コントロールボックスに接続されたデータ処理用PCにて生画像が自動的に処理され、空間ゲノミクス測定値をすぐにダウンストリーム解析に使用できる形式で出力します。
- 検出された転写産物のリスト (.CSV、.PARQUET)
- モザイク画像 (.TIFF)
- 細胞マトリックスごとの転写産物 (.CSV)
- 細胞メタデータ (.CSV)
- 細胞境界 (.PARQUET)
- 細胞クラスタリングアノテーション (.CSV、.H5AD)
* Spatially resolved, highly multiplexed RNA profiling in single cells. Science. Chen KH, Boettiger AN, Moffitt JR, Wang S, Zhuang X. 2015;348(6233):aaa6090-aaa6090
MERFISHは、シングルセルゲノミクスにおける画期的な技術であり、基礎科学から創薬、臨床病理学まで、基礎生物学と医学の両分野で幅広く応用されています。MERFISH は、細胞本来の状態を保ちながら幅広い存在量の RNA を定量できるため、培養中の個々の細胞や複雑な組織の in situ トランスクリプトーム解析の多くのアプリケーションを可能にします。
MERSCOPE タンパク質染色キットによって、RNA転写産物とタンパク質の同時検出が行えるようになり、空間マルチオミクス測定も可能になりました。
アプリケーション事例
 正常およびアルツハイマー病ヒト大脳皮質の分子的・空間的不均一性の同時プロファイリング 正常およびアルツハイマー病ヒト大脳皮質の分子的・空間的不均一性の同時プロファイリング |
正常およびアルツハイマー病 (AD) 患者の凍結保存ヒト脳ブロックの切片を、MERSCOPE Largeスライド上にマウントしました。Largeスライドに対してMERFISH 2.0のサンプル調製ワークフローを実施し、細胞タイピングおよび神経変性経路のマーカーを含む960plex の遺伝子パネルを用いた空間情報のプロファイリングを行いました。同一実験内で正常およびアルツハイマー病の脳を解析することで、疾患状態における細胞タイプ特異的な分子および細胞の局在を特定することが可能になりました。
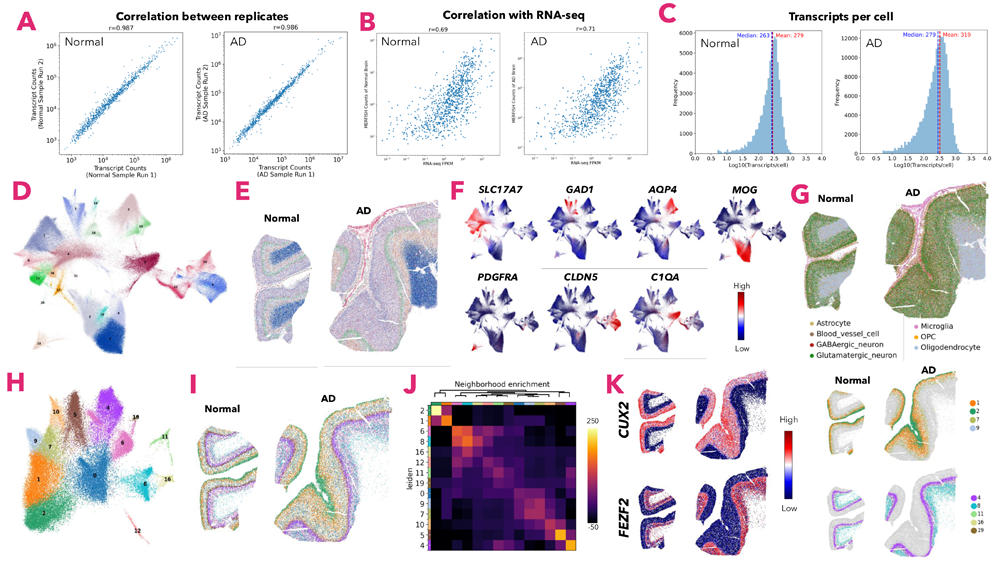
Figure: (A) 960plex遺伝子パネルを用いた正常およびAD脳スライスでのMERSCOPE replicate測定からの転写産物数の相関を示す散布図。(B) 正常またはADヒト大脳皮質の転写産物数と同じ組織のバルクRNA-seqとの相関を示す散布図。(C) 1回のMERSCOPE Ultra実験における正常およびAD脳サンプルの細胞あたりの転写産物数の分布を示すヒストグラム。(D) 正常およびADヒト大脳皮質切片からのMERSCOPEデータの教師なしクラスタリング解析から得られた細胞クラスターを示すUMAP。(E) 正常およびADのヒト大脳皮質における細胞クラスターの空間分布。(F) 全細胞における主要な細胞集団 マーカー遺伝子の発現レベルを示すUMAP。(G) ヒト大脳皮質における主要な細胞タイプの空間分布。(H) グルタミン酸作動性ニューロンサブクラスターのUMAPと (I) 空間分布。(J) 異なるグルタミン酸作動性サブクラスターの近傍エンリッチメントを示すヒートマップ。(K) (左) ヒト大脳皮質におけるCUX2とFEZF2の異なる発現パターンを示す空間ヒートマップ。(右) CUX2 (上) またはFEZF2 (下) を発現する異なるグルタミン酸作動性サブクラスターの空間分布。
Vizgen 社 SFN 2024 ポスター“Advances in Spatial Transcriptomics: Using 1000-plex Panels to Unlock Insights from Brain Tissue Samples with MERSCOPE® Ultra and MERFISH 2.0” より引用
▲ アプリケーション事例一覧へ戻る
 高感度MERSCOPEワークフローによる腫瘍微小環境中の細胞タイプのプロファイリング 高感度MERSCOPEワークフローによる腫瘍微小環境中の細胞タイプのプロファイリング |
MERFISH 2.0ワークフローにより、FFPE子宮がん組織の腫瘍微小環境中の細胞タイプを詳細にプロファイリングすることが可能です。腫瘍サンプル中の主要なクラスターに含まれる免疫細胞のマーカーを用いて、UMAPと空間解析により組織全体の遺伝子数を再クラスター化することができます。

Figure : (A) FFPE 子宮がん組織とその結果得られたLeidenクラスターを空間的に、またUMAP上にプロットした。(B) 各Leidenクラスターにおける上位の差次的発現遺伝子をドットプロットを用いて可視化した。(C) 細胞クラスターを、確立されたマーカー遺伝子に基づいて主要な細胞集団にグループ化した。(D) 全細胞における主要な細胞集団マーカー遺伝子の発現レベルを示すUMAP。(E) 主要な細胞集団の空間分布を、(C) と同様に細胞グループごとに色分けしてプロットした。(F) (B) のドットプロットのクラスター6、10、9、5 に含まれている免疫細胞のマーカーを用いて、UMAPと空間解析により組織全体の遺伝子数を再クラスター化し、T細胞、末梢性T細胞、細胞傷害性T細胞、ヘルパーT細胞、B細胞等の免疫細胞に分類することができた。いくつかの免疫細胞タイプの遺伝子発現をドットプロット、UMAP、空間分布にプロットした。
Vizgen 社 AACR2024 ポスター “Improved spatially resolved single cell transcriptomic imaging in archival tissues with MERSCOPE®” より引用
▲ アプリケーション事例一覧へ戻る
 マウス脳受容体のマッピング マウス脳受容体のマッピング |
 脳内に存在する約370の非感覚的GPCRのうち約90%がシグナル伝達を担い、脳の老化や神経変性疾患において重要な役割を果たしていると考えられていますが、その構造特性、存在量の少なさ、特異性の高い抗体の不足により解析が困難です。
脳内に存在する約370の非感覚的GPCRのうち約90%がシグナル伝達を担い、脳の老化や神経変性疾患において重要な役割を果たしていると考えられていますが、その構造特性、存在量の少なさ、特異性の高い抗体の不足により解析が困難です。
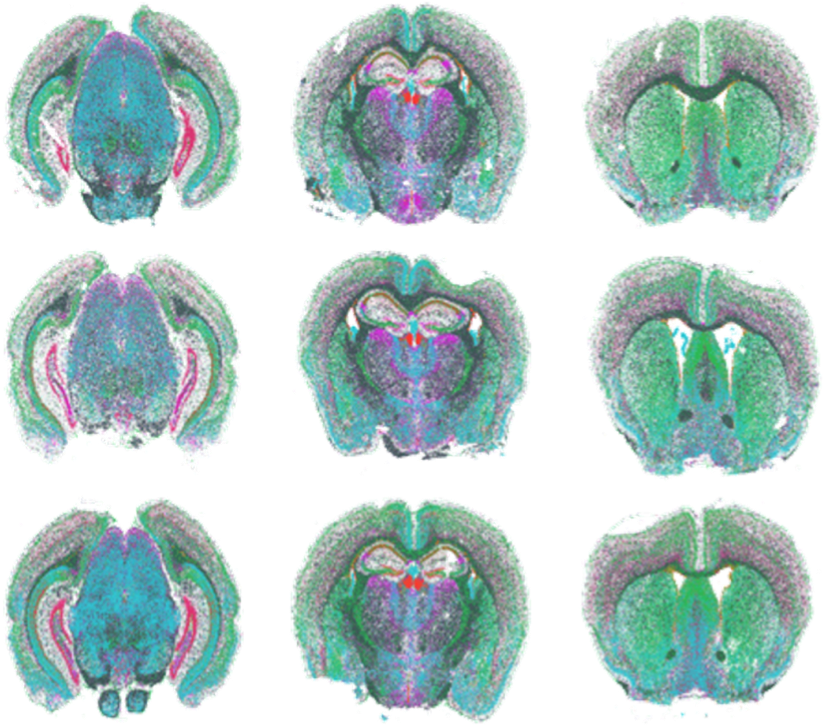
MERFISH Mouse Brain Receptor Mapを開発するために、Vizgenは、標準的な脳細胞型マーカー、非感覚性GPCR、受容体型チロシンキナーゼを含む483個の遺伝子からなるパネルを構築し、マウス脳冠状スライスを用いて、細胞背景とともに脳全体の非感覚性GPCR発現の空間的プロファイルを作成しました。MERFISH測定は、脳の3つの位置の完全冠状切片について、各位置について3つの生物学的replicateを用いて行われ、標的転写産物の正確な位置を決定しました。
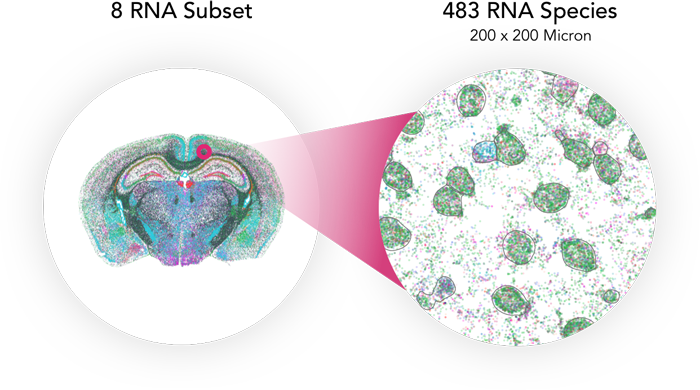
Figure 1:MERFISH測定による483の転写産物からの8つのRNAサブセット。
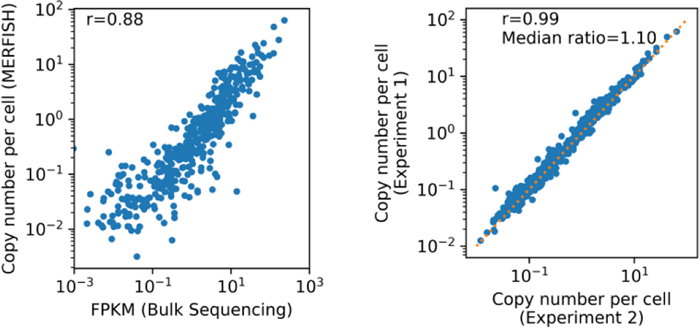
Figure 2:検出された転写産物の数をバルクシーケンシングと比較したところ、強い相関性(r=0.88)が見られ、生物学的replicate間にも強い相関性が見られた。
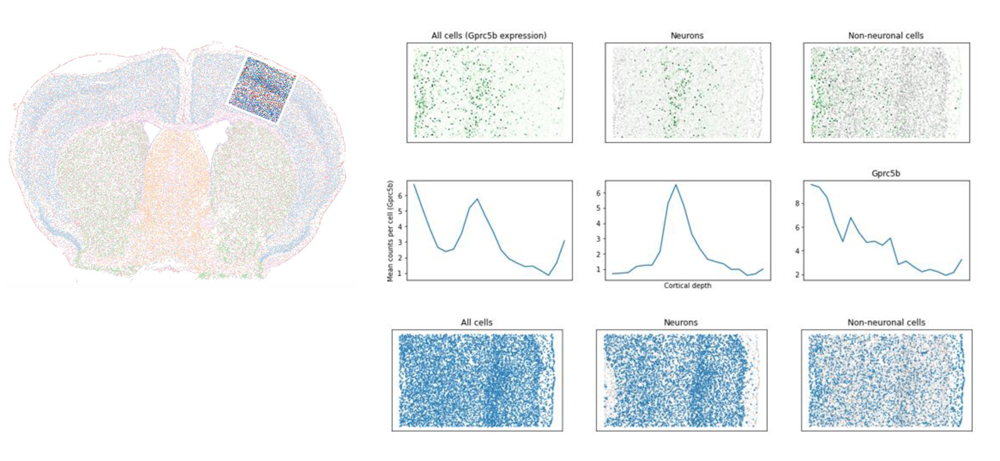
Figure 3:細胞タイプに依存したGprc5b発現の空間分布。大脳皮質の深部と中部においてGprc5bの高発現のピークが見られる。神経細胞と非神経細胞のシグナルのデコンボリューションにより、大脳皮質中部のピークは神経細胞内の発現が支配的であり、大脳皮質深部のGprc5高発現のピークは非神経細胞内の発現が支配的であることが判明した。このように、MERFISHではシングルセルの分解能で、遺伝子発現の空間的、細胞タイプ依存的な差異をマッピングすることが可能である。
Vizgen社 HCA Poster 2021: MERSCOPE Reveals the Transcriptional Organization of the Mouse Brain より引用
▲ アプリケーション事例一覧へ戻る
 大腸がん細胞と微小環境の新たな空間的関係 大腸がん細胞と微小環境の新たな空間的関係 |
腫瘍微小環境 (TME) を理解するためには、単に細胞の種類や遺伝子プログラムのカタログだけでは不十分であり、細胞の空間的な構成や、どこで多細胞相互作用ネットワークを形成しているかを見ることが重要です。
この事例では、ヒトのミスマッチ修復機能が低下している MMRd 大腸がん (CRC) と機能が保たれている MMRp 大腸がん標本の、シングルセル空間分解トランスクリプトーム解析を行いました。
腫瘍の変異負荷が高いMMRd腫瘍は、MMRp腫瘍と比較して高い細胞溶解性T細胞浸潤を特徴とする免疫応答を有することが知られており、シングルセルの空間プロファイリングと免疫駆動プログラムがこれらの腫瘍間でいかに異なるかを理解するのに理想的なシステムです。
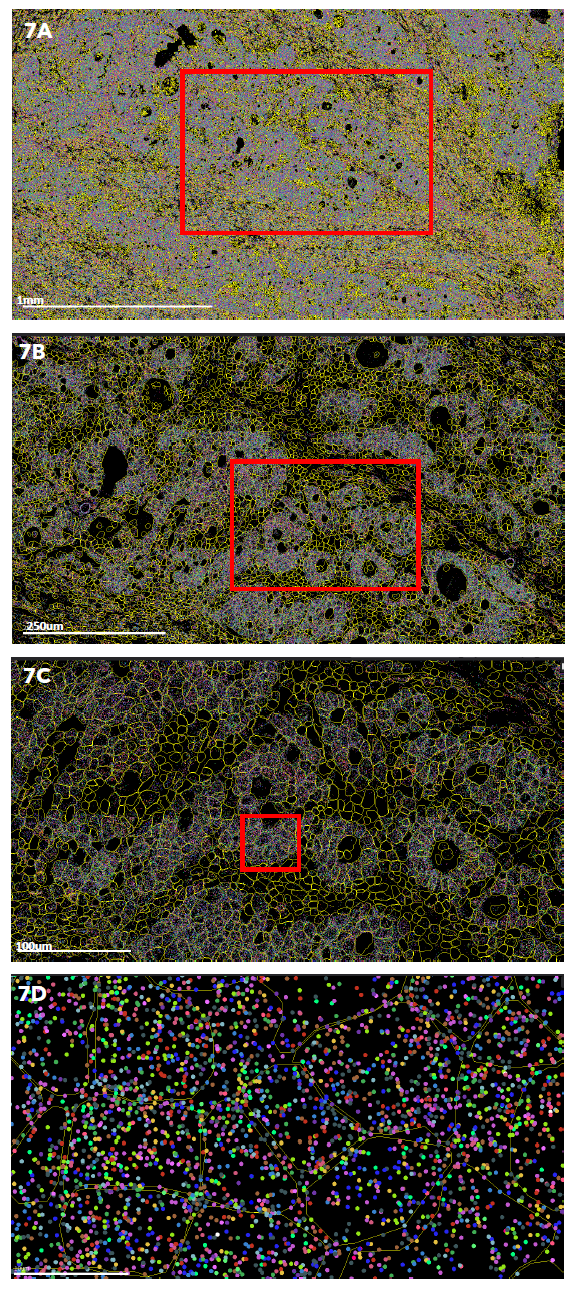 |
Figure: FFPE結腸がん患者サンプルにおけるMERFISHデータをMERSCOPE Vizualizerで可視化したもの。 データは組織全体 (7A) から個々の細胞 (7D) まで様々な倍率で表示され、細胞のセグメンテーション (黄色) とすべてのRNA転写物の発現が表示されている。
このデータからUMAP※細胞クラスターを生成し (7E)、それらのクラスターを組織上に投影して、各細胞タイプの位置を特定することができる (7F)。 細胞のセグメンテーションを行うために、社内で開発した細胞境界染色をDAPI (data not shown) と組み合わせて使用した。これらの細胞の種類と位置を特定することで、既知および新規の細胞間相互作用が明らかになり、がん細胞と微小環境の関係がより明確になる (7F)。
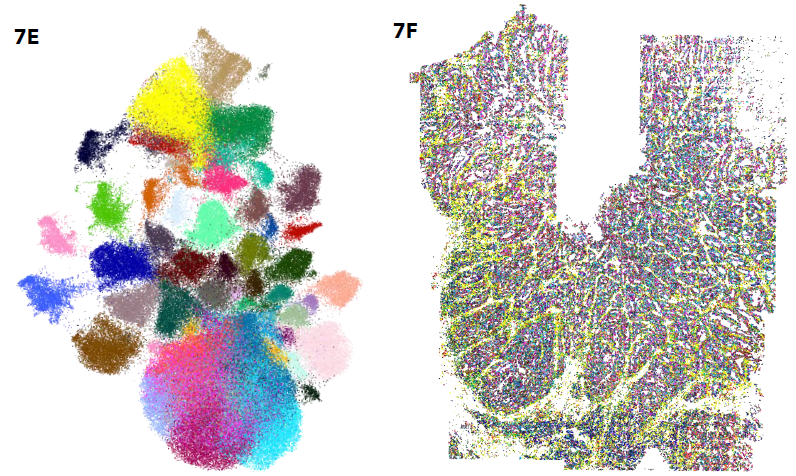
※UMAP (Uniform Manifold Approximation and Projection、一様多様体近似と投影)
|

がん細胞と腫瘍微小環境のクラスターを、空間的に関連したアルファ近傍に構築した。細胞のクラスターが特定されたら、0.65ミクロンのシェイプを定義して、各クラスターのアルファシェイプを構築した。 互いにその距離内にある細胞を近傍として定義し、統計的に有意かどうかを判断した (8A)。 その結果、がん細胞と微小環境細胞の相互作用やシグナル伝達はランダムではなく、その構成が生物学的に関連していることが示唆された (8B)。
Vizgen社 AACR 2022 Poster : A single-cell spatially resolved map of colorectal cancer identifies novel spatial relationships between cancer cells and the microenvironment より引用
▲ アプリケーション事例一覧へ戻る
 ヒト大腸がんの腫瘍微小環境における免疫細胞のマッピング ヒト大腸がんの腫瘍微小環境における免疫細胞のマッピング |
ヒトの大腸がんの腫瘍微小環境において、417遺伝子のプロファイリングを行いました。 左の図は、この417種類の遺伝子のうち、10種類のRNAサブセットの空間分布を表しています。
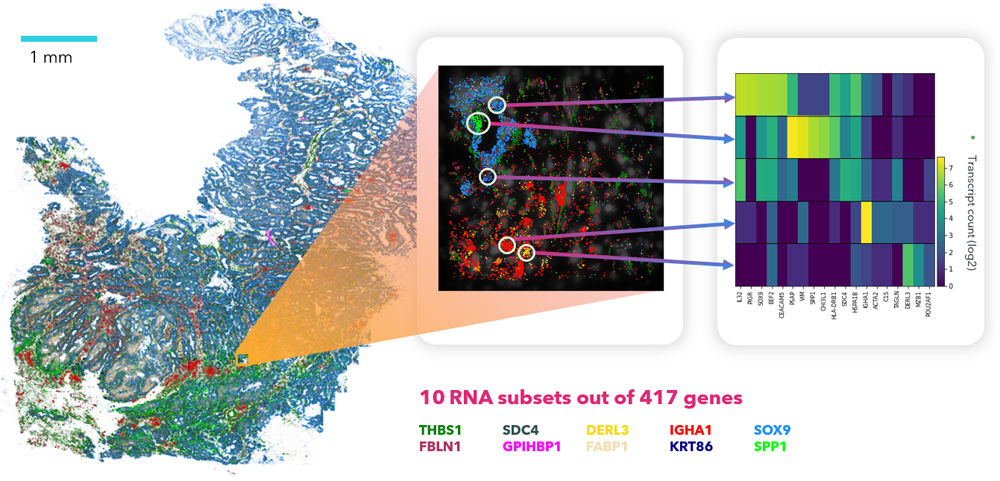
このTranscriptの情報から、細胞クラスタリング解析を行った結果、B細胞、CD8 positive T細胞、NK細胞、マクロファージといった、組織内で転写的に異なる細胞タイプを同定することが出来ました。
中央の拡大図において、青いドットはSOX9+の腫瘍細胞群、この腫瘍細胞群に囲まれた緑色のドットはSPP1発現ストローマ細胞を示しています。赤いドットは免疫グロブリンα1発現で示される、B細胞の大きな集団です。個々の細胞に焦点を当て、その細胞に発現している遺伝子をヒートマップ表示させることも可能です。
▲ アプリケーション事例一覧へ戻る
 神経膠腫と微小環境の細胞間相互作用の解析 神経膠腫と微小環境の細胞間相互作用の解析 |
この研究では、微小環境下における神経膠芽腫、特にグリオブラストーマ細胞と免疫細胞の相互作用を調査しました。筆者らは、MERFISHを利用して、マクロファージが濃縮されている可能性が高い腫瘍サンプルの領域で、135種類の遺伝子プロファイルを解析しました。
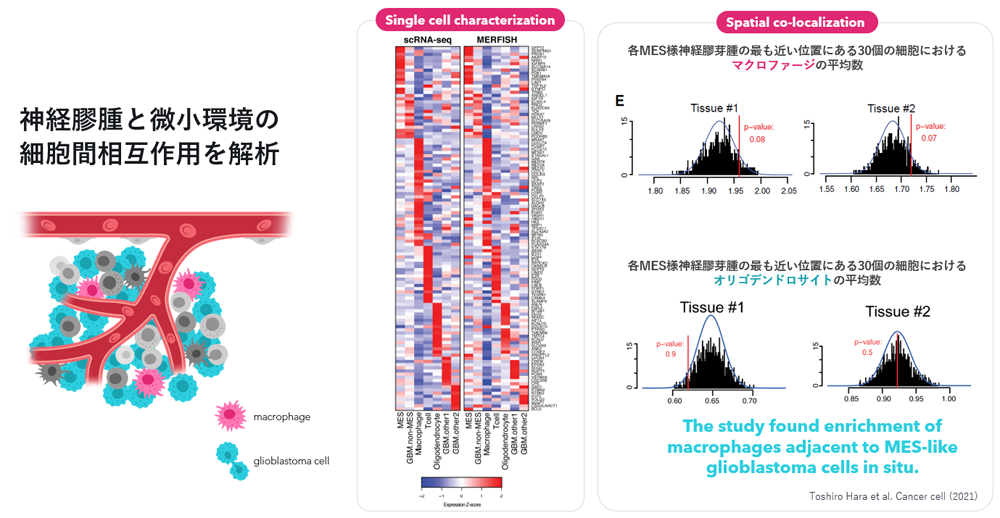
ヒートマップは、ヒトのグリオブラストーマ組織切片における、遺伝子のプロファイリング結果です。RNAseqとMERFISHのプロファイリングで、両者の間には相関係数0.82の、高い相関性が見られました。
さらに、間葉系ライクな神経膠芽腫の周辺において、マクロファージが特異的に濃縮されていることを発見しました。右上の図は、間葉系ライクな神経膠芽腫を中心としたときに、その近傍にある最も近い30個の細胞の中に含まれるマクロファージの平均数を表したヒストグラムです。ランダムに予想される分布を黒のバーで、実際に観測された平均値を赤線で示しています。Tissue #1、Tissue #2いずれも、黒の分布のピークよりも右側に実際の観測平均値が位置しており、間葉系ライクな神経膠芽腫の周辺には、マクロファージが有意に多く存在していることが示されました。
一方で、オリゴデンドロサイトで行った同様の解析 (右下) では、マクロファージの有意な濃縮・局在は見られないことが示されました。その後、筆者らは、マウスモデルで実験を続け、マクロファージがin vitroおよびin vivoで間葉系ライクな神経膠芽腫の状態を誘導することを実証しています。
▲ アプリケーション事例一覧へ戻る
 マウス視床下部の155種転写産物の解析 マウス視床下部の155種転写産物の解析 |
MERFISHを用いて、マウス視床下部の黒枠 (Fig.1、左) で囲った領域における50万の細胞について、155種類の遺伝子プロファイルを作成しました。
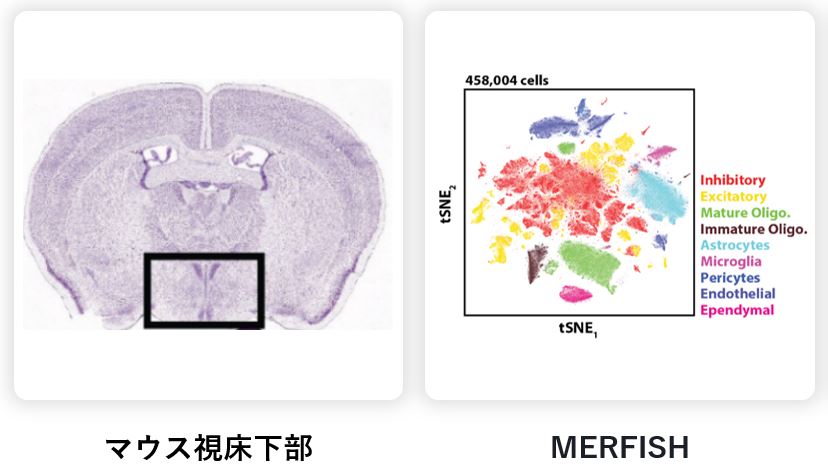
Figure 1: (右)細胞間の遺伝子発現プロファイル情報から、細胞クラスタリングを行ったt-SNEプロット。このプロットから、この脳領域では75個の細胞クラスターが同定され、興奮性ニューロンと、抑制性ニューロンで異なる細胞クラスターを確認することができた。
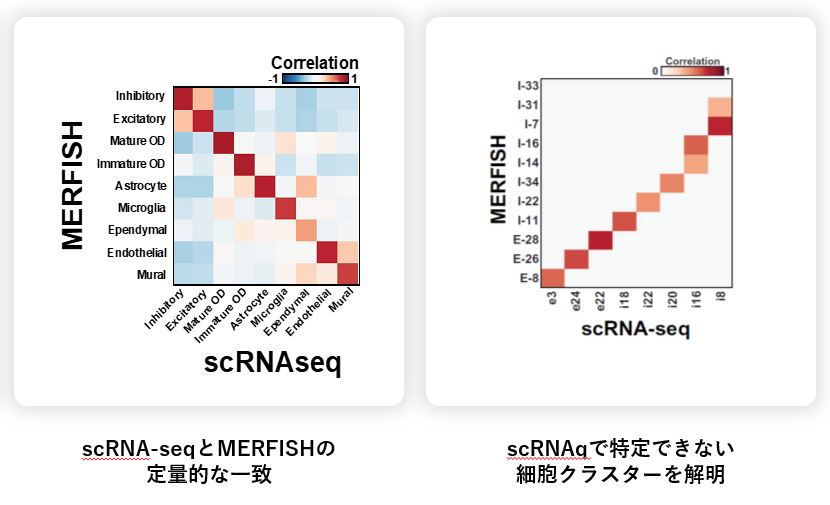
Figure 2:(左) scRNAseqによって見出されたすべての主要な細胞型は、MERFISH測定によっても同様に再現された。(右) scRNAseq では 1 つのクラスターとして表示される右上の 2 つの細胞クラスターが、MERFISHの解析では2つに分離できた。これらは、MERFISHの高い感度と、高い細胞スループットにより実現した典型的な例だと言える。
Reference: Molecular, spatial, and functional single-cell profiling of the hypothalamic preoptic region (

)
Jeffrey R Moffitt et al., Science. 2018 Nov 16;362(6416):eaau5324. doi: 10.1126/science.aau5324. Epub 2018 Nov 1.
▲ アプリケーション事例一覧へ戻る
 マウス視床下部視索前野における細胞機能の解明 マウス視床下部視索前野における細胞機能の解明 |
視床下部の視索前野は、多くの社会的行動やホメオスタシスを制御しています。 マウスの視床下部を前後軸に沿って60枚の等間隔スライスに切り出し、5枚目のスライスごとにMERFISH測定を行い、組織内の三次元的な細胞構成を評価しました。
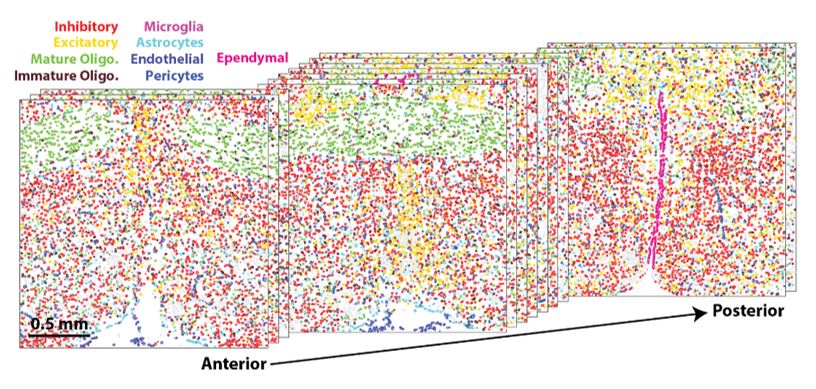
Figure 1:MERFISHを用いて脳組織内の、約110万の細胞を解析した。その結果、視床下部には驚くほど多様なニューロンが存在し、その多くはこれまで知られていなかった約70の異なるニューロン集団から構成されていることを明らかにした。ドットは主要な細胞クラスの種類と位置を示す(左側が脳の前方、右側が脳の後方)。
例えば抑制性のニューロン集団は、視索前野でより多く存在している一方で、興奮性ニューロンはいくつかの領域で特異的に濃縮されていたものの、脳の後方に行くほど分散するような分布をしていることが画像的に解析できた。
さらに、子育て、交尾、攻撃といった社会的な行動によって活性化される、特定のニューロン集団を同定しました。Fos遺伝子は、神経の活動電位の発火の際にしばしば発現するため、Fosの発現は神経活動の間接的なマーカーとして利用されます。各ニューロン集団における、FOS遺伝子の発現を測定しました。
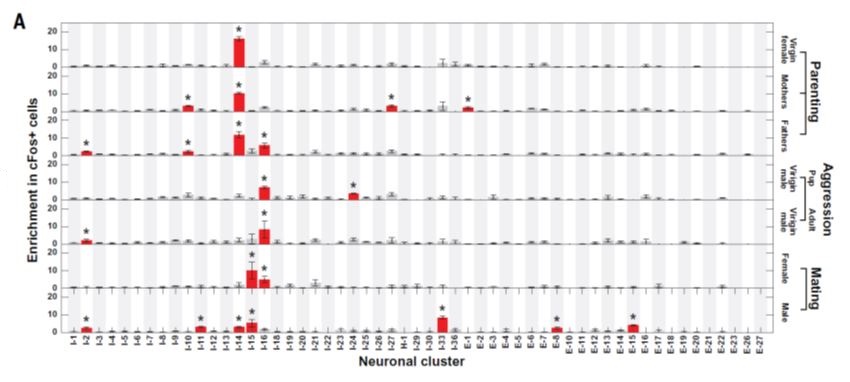
Figure 2:横軸はニューロン細胞のクラス分けを示し、赤いバーはFosの発現量 (ニューロン集団の活性度)を反映している。右側には、交尾、子育て、攻撃といった社会的行動のカテゴリを示す。特定のニューロン集団が選択的に活性化されている。
これらの結果から、転写的に異なるニューロンタイプが視床下部の特定の機能を制御している、という考えが支持されました。
また、子育てをする全ての動物で活性化される、中核的な神経細胞集団と、母親と父親で異なる活性化を示す細胞集団を同定し、生理状態が子育て行動にどのように影響するかを明らかにしました。さらに、オスとメスの性行動や、オスによる攻撃性に関連する細胞も同定されました。
これらの結果は、行動回路のメカニズムを研究するための枠組みを提供し、将来的には、バイオマーカーの開発、開発候補品の作用機序研究へとつながる、強力なツールになると言えます。
Reference: Molecular, spatial, and functional single-cell profiling of the hypothalamic preoptic region (

)
Jeffrey R Moffitt et al., Science. 2018 Nov 16;362(6416):eaau5324. doi: 10.1126/science.aau5324. Epub 2018 Nov 1.
▲ アプリケーション事例一覧へ戻る
 神経細胞の位置特異的な転写産物の同定 神経細胞の位置特異的な転写産物の同定 |
神経細胞は複雑な神経突起の形態を持つ、高度に偏向した細胞です。樹状突起と軸索における転写産物の空間的な局在は、神経細胞の機能において重要な役割を担っています。 この研究では、MERFISHを用いて、個々の神経細胞内の転写産物のプロファイリングを行い、サブマイクロメートルからミリメートルまでの、複数の長さスケールにわたって千種類を超えるRNAの空間構成をマッピングしました。
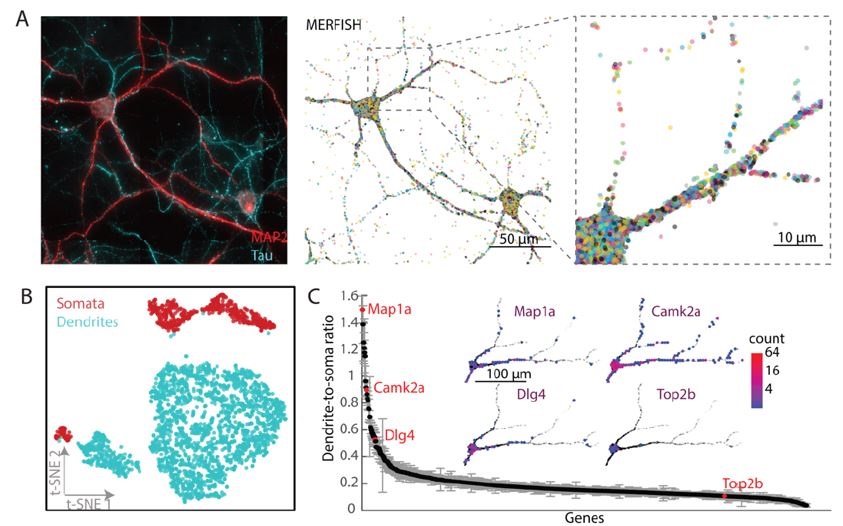
Figure:(A・左) 4mm × 4mmの領域における、MAP2とTauの免疫染色画像。(A・右) 同じ領域の転写産物。 軸索、樹状突起といった異なる神経細胞区画におけるRNAを、直接可視化できていることが示されている。(B) トランスクリプトームプロファイルに基づいた、興奮性ニューロンと抑制性ニューロンの、体細胞および樹状突起におけるtSNEプロット。(C) 950遺伝子の測定において、すべての注釈付きニューロンで測定された個々のRNA種の樹状突起-体細胞比を、RNA種 (遺伝子) の樹状突起-体細胞比に基づいてランク付けした。エラーバーは4つの生物学的replicateから決定されたSEMを表す。(C・挿入図) 1つのニューロンにおける、赤色でハイライトされた4つのRNA種の空間マップ。
この解析によって、神経細胞内のトランスクリプトームの細胞内アトラスを作成し、軸索と樹状突起においてRNAの濃縮が起きていること、そして、樹状突起における多様なRNA分布パターンを明らかにしました。
さらに、RNAのクラスター形成が、タンパク質合成および、シナプス活性に依存していることなど、サブマイクロメートルの空間的な関連性を持つRNA種を同定しました。
これらの結果は、神経細胞におけるRNAの局在と、局所的なRNAの機能に関する、定量的な研究基盤を提供するものだと言えます。
Reference: Spatial organization of the transcriptome in individual neurons (

)
Guiping Wang et al., BIORXIV DOI:10.1101/2020.12.07.414060 (2020)
▲ アプリケーション事例一覧へ戻る
 FFPEマウス脳切片の in situ シングルセルトランスクリプトーム・イメージング FFPEマウス脳切片の in situ シングルセルトランスクリプトーム・イメージング |
FFPE サンプルにおいて MERFISH実験が可能であることを実証するため、483 種類のマウス脳受容体遺伝子パネルを使用して、FFPE マウス脳をイメージングしました。MERFISHのカウントをバルクRNA-seqと比較し、この測定が高感度かつ正確であることが確認されました。 さらに、MERSCOPE FFPE ワークフローの感度と精度を評価するために、新鮮な凍結サンプルから得られた MERFISH 結果と、一致する脳領域から得られた FFPE サンプルからのデータを比較しました。両者の各細胞クラスターは同様の空間分布を示し、作製方法の異なるサンプルでも正確に転写産物の測定が可能であることが分かりました。
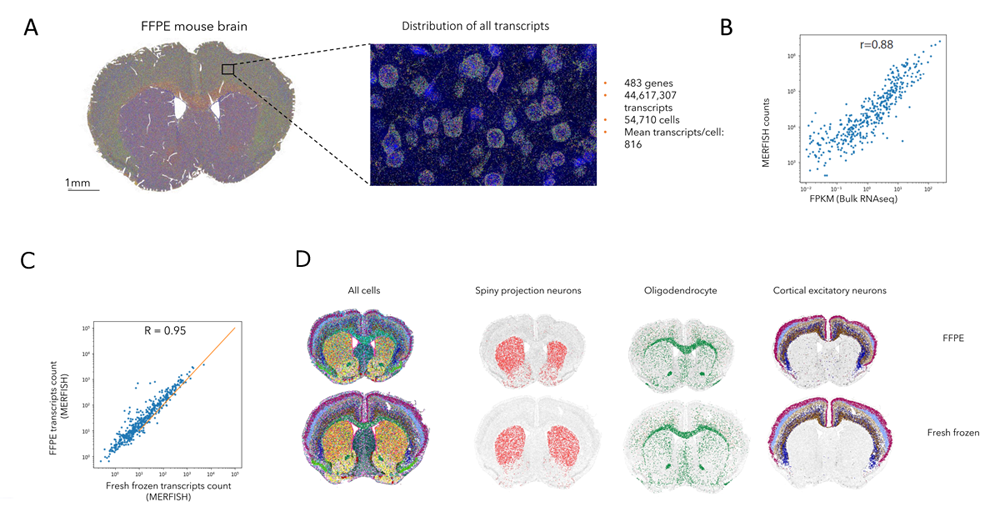
Figure:A) FFPEマウス脳を483種類のマウス脳受容体パネルを用いてMERSCOPEでイメージングした。すべての転写産物の空間分布を、脳の冠状断面(左)およびDAPIオーバーレイを伴う拡大した皮質領域(右)にわたって示す (スケールバ:1mm)。B) MERSCOPEカウントとバルクRNAシーケンスデータの相関から、この測定が高感度かつ正確であることがわかる。C) マウス脳のFFPEと新鮮凍結切片の転写産物量の相関性評価。非常に高い相関性を示す。D) FFPEマウス脳 (上) と新鮮凍結マウス脳 (下) で同定された細胞タイプの空間分布。
Vizgen社ポスター ”In Situ Single-cell Transcriptomic Imaging in Formalin-fixed Paraffin-embedded Tissues with MERSCOPE™” より引用
▲ アプリケーション事例一覧へ戻る
 タンパク質の共検出により、RNA転写産物では捉えられない複雑な細胞構造を明らかにする タンパク質の共検出により、RNA転写産物では捉えられない複雑な細胞構造を明らかにする |
MERSCOPE タンパク質染色キットを使⽤することで、RNA転写産物と同時に最⼤6種類 (細胞境界染色を行う場合は最大5種類) のタンパク質を検出することができます。
マウスの脳組織スライスを、6種類のタンパク質︓神経細胞核マーカー NeuN、ミクログリアマーカー Iba1、オリゴデンドロサイトおよびミエリン形成マーカー MOGおよび MBP、内⽪細胞マーカー CD31、アストロサイトグリアマーカー GFAPに対する抗体、および対応するMERSCOPE タンパク質染色キットを使⽤して染色しました。MERFISHイメージング⽤には468種類の遺伝⼦パネルを選択しました。
MERSCOPEはマウス脳内の6種類のタンパク質と468種類の遺伝⼦をすべて検出することに成功しました (Fig.AおよびFig.B)。ピアソンの相関係数を用いたMERFISHカウントとバルクRNAシーケンスデータの関係評価からr=0.83という強い相関が得られ (Fig.C)、MERFISHのデータが⾮常に正確であることが⽰されました。これとは別に、MERFISHカウントはタンパク質染⾊の有無にかかわらず、相関係数が1前後、⽐が0.89と強い相関を維持しており、タンパク質共染⾊がMERFISHイメージングに影響を与えないことが⽰唆されました (Fig.D)
タンパク質の共染⾊を含めることで、MERSCOPE は主要な細胞マーカーの細胞形態を明らかにすることができます。例えば、海⾺領域では、CA1亜領域の特徴的な錐体細胞はNeuN+細胞によって縁取られ (Fig. 1E)、MBP+の有髄の構造は脳梁領域内に存在していました (Fig. 1K)。
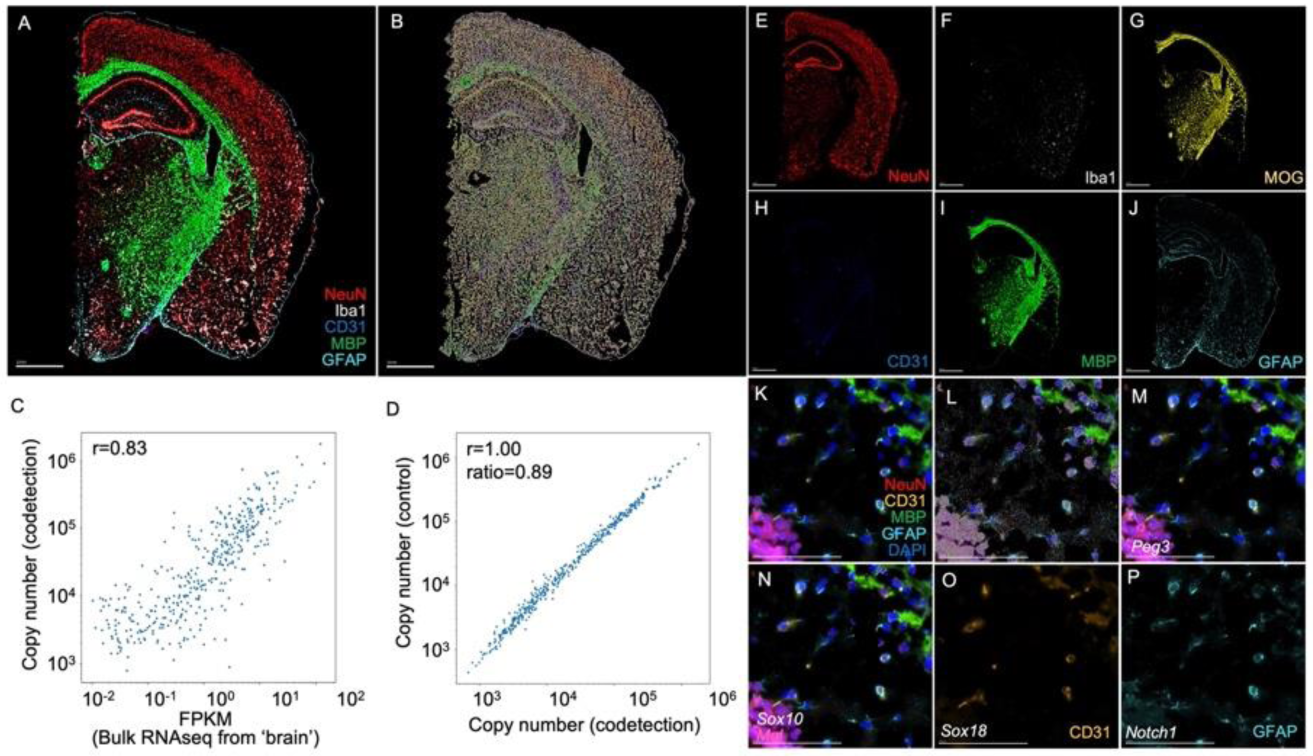
Figure:MERSCOPEによるマウス脳内の6種類のタンパク質と468種類の遺伝⼦の共検出。A) 5種類のタンパク質染⾊のマージ画像:NeuN-⾚、Iba1-⽩、CD31-⻘、MBP-緑、GFAP-⽔⾊、B) マウス脳において、タンパク質染⾊は検出されたすべてのRNA転写産物と重なっている、C) MERFISHデータとマウス脳由来のバルクRNAシーケンスデータとの相関解析、D) タンパク質共検出を⾏った、または⾏っていないサンプルからのMERFISHデータの相関、E-J) 各個別のタンパク質の免疫染⾊︓NeuN-⾚ (E)、Iba1-⽩ (F)、MOG-⻩ (G)、CD31-⻘ (H)、MBP-緑 (I)、GFAP-⽔⾊ (J)、K) 海⾺ CA1-脳梁領域のタンパク質共染⾊ (NeuN-⾚、CD31-⻩、MBP-緑、GFAP-⽔⾊) とDAPI (⻘) 染⾊、L) Kで⽰したのと同じ領域でMERFISHによって検出したすべての転写産物の空間的な分布、M) 4種類のタンパク質共染⾊と重ね合わせた遺伝⼦Peg3の空間分布、N) 4種類のタンパク質共染⾊と重ね合わせた遺伝⼦Sox10/Malの空間分布、O) 単⼀のタンパク質CD31とマージした遺伝⼦Sox18の空間分布、P) 単⼀のタンパク質GFAPとマージした遺伝⼦Notch1の空間分布。
Vizgen社 Application Note "Protein Co-Detection with MERSCOPE™"より引用
▲ アプリケーション事例一覧へ戻る
 タンパク質の共検出により、シングルセル解析における細胞タイプのアノテーションを⽀援する タンパク質の共検出により、シングルセル解析における細胞タイプのアノテーションを⽀援する |
Vizgenは、不均⼀性の⾼いがんサンプルを⽤いて、MERSCOPEタンパク質染色キットが細胞タイプまたは状態のアノテーションに役⽴つかどうかを検証しました。ヒト⼤腸がん組織においてタンパク質染色キットと241種類の遺伝⼦のMERFISHパネルによる5-plexタンパク質共染⾊を、細胞のセグメンテーションと下流のシングルセル解析を容易にするための細胞境界染⾊と共に⾏いました (Fig.AおよびB)。
タンパク質染⾊を⾏わなかったサンプルでは、FOV (200μm X 200μm) あたり平均42,656の転写産物カウントが検出され、タンパク質共染⾊を⾏ったサンプルでは、FOVあたり35,441遺伝⼦カウントが検出されました (Fig.C)。ピアソンの相関係数から、MERFISHの結果はこれら2つの条件の間で r=0.97 と⾼い相関があり、MERFISHによるタンパク質-転写産物共検出の測定は信頼性が⾼いことが⽰されました (Fig.D)。panCK、VIM、Tubulin、HER2、KI67 の⾼解像度画像は、サンプル内で予想される空間パターンを⽰し (Fig. E-I)、MERSCOPE タンパク質検出キットが適⽤できることが確認できました。
MERSCOPEはマウス脳内の6種類のタンパク質と468種類の遺伝⼦をすべて検出することに成功しました (Fig.AおよびFig.B)。ピアソンの相関係数を用いたMERFISHカウントとバルクRNAシーケンスデータの関係評価からr=0.83という強い相関が得られ (Fig.C)、MERFISHのデータが⾮常に正確であることが⽰されました。これとは別に、MERFISHカウントはタンパク質染⾊の有無にかかわらず、相関係数が1前後、⽐が0.89と強い相関を維持しており、タンパク質共染⾊がMERFISHイメージングに影響を与えないことが⽰唆されました (Fig.D)
細胞境界染⾊を⽤いて細胞の境界を明確にすることで、腫瘍内の個々の細胞をセグメント化し、下流のシングルセル解析を⾏うことができました (Fig. J)。シングルセルのクラスタリング解析により、UMAPに10個のユニークな細胞クラスターが⾒つかり (Fig. O)、また、同定された各細胞の空間座標を登録し、組織全体の各細胞タイプの空間分布をマッピングしました (Fig.P)。タンパク質の共染⾊情報を使って、腫瘍内の細胞タイプをさらにアノテーションすることができました (Fig. Q-T)。例えば、Leiden-0は、PLA2G2A、ASCL2、LDHBのようないくつかの重要ながんマーカー遺伝⼦の発現を伴ったpanCKポジティブのタンパク質染⾊に基づき、がん細胞のサブタイプであるように⾒えました (Fig. Q)。
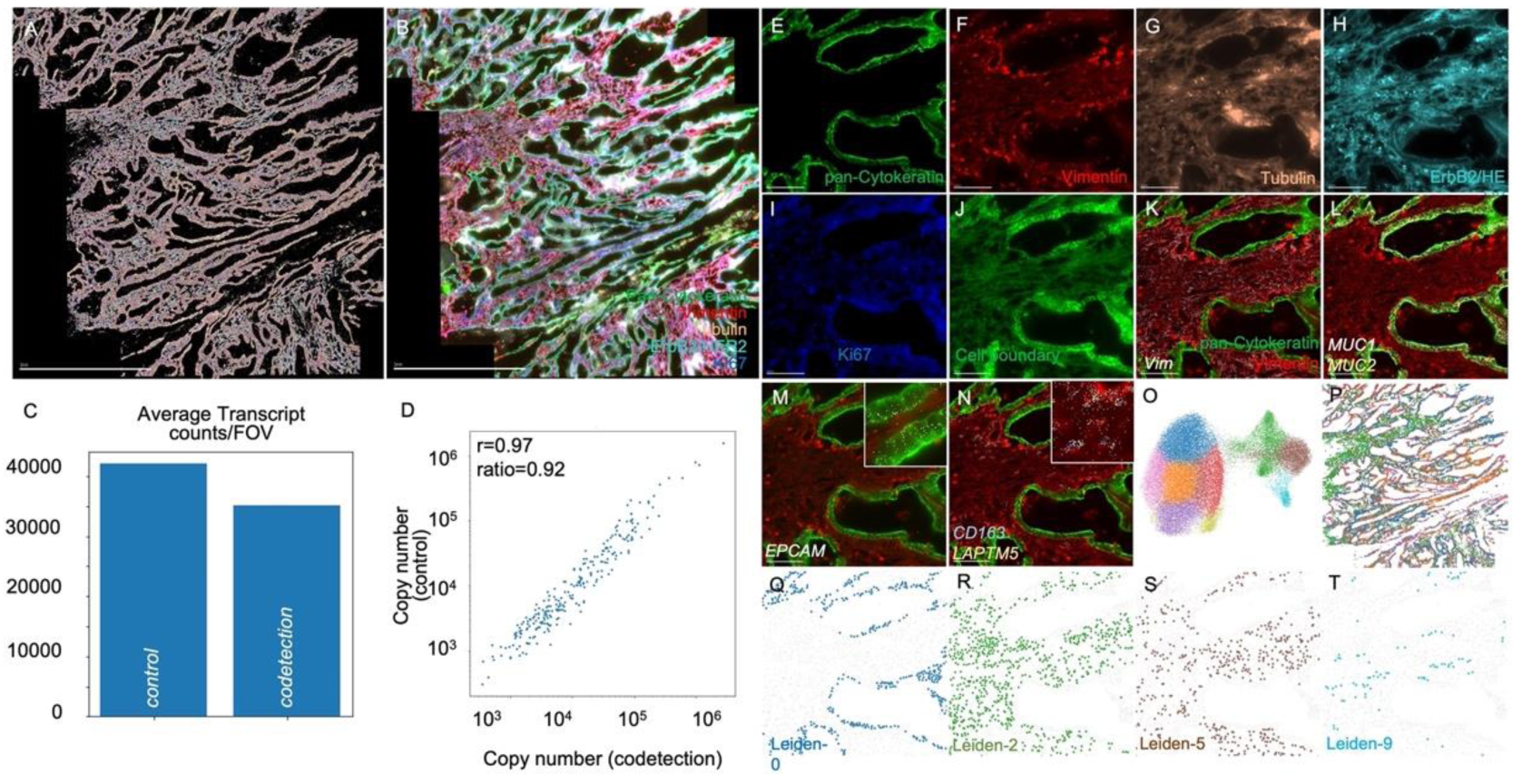
Vizgen社 Application Note "Protein Co-Detection with MERSCOPE™"より引用
▲ アプリケーション事例一覧へ戻る
 FFPEヒト腫瘍のシングルセル空間分解トランスクリプトーム・プロファイリング FFPEヒト腫瘍のシングルセル空間分解トランスクリプトーム・プロファイリング |
MERFISHにより、500遺伝子のパネルを用いて8種類のFFPEヒト腫瘍の細胞アトラス作成と免疫プロファイリングを行いました。各サンプルで数十万個の細胞が画像化され、1サンプルあたり数億カウントが生成されました。MERSCOPE FFPE Immuno-oncology Data Releaseにより、免疫細胞のサブタイプを含む各腫瘍の主要な細胞タイプをすべてマッピングし、それらの遺伝子発現プロファイルをシングルセルの分解能で特徴付けることができました。
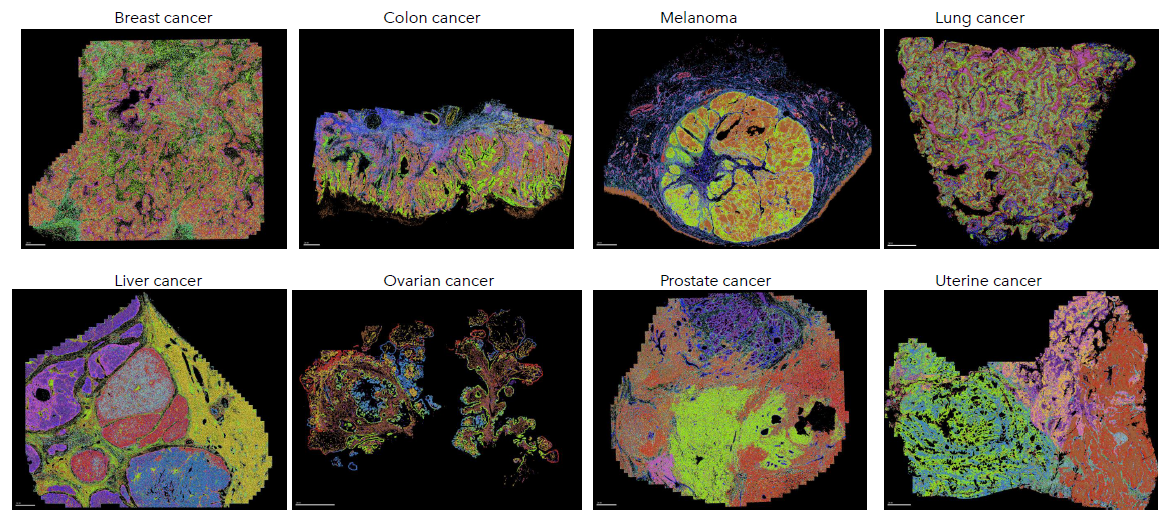
FFPEヒトがんサンプルにおいて識別された細胞タイプの空間分布。乳がん、大腸がん、メラノーマ、肺がん、肝臓がん、卵巣がん、前立腺がん、子宮がんのFFPEサンプルにおいて、500種類の遺伝子パネルを用いてMERSCOPEで解析したもの。合計40億以上の転写産物、900万個の細胞を検出。細胞はセグメント化、シングルセル解析が行われ、各サンプルで識別された細胞は、サンプル内での異なる細胞の空間分布を示すために色分けされている。スケールバー : 1 mm
※本データはFFPE Human Immuno-Oncology Data Release としてダウンロード可能です。
※本実験で使用した遺伝子パネルは、「Immuno-Oncologyプレデザイン遺伝子パネル」としてご注文いただけます。
Vizgen社ポスター ”In Situ Single-cell Transcriptomic Imaging in Formalin-fixed Paraffin-embedded Tissues with MERSCOPE™” より引用
▲ アプリケーション事例一覧へ戻る
【 神経科学 】
 The contribution of de novo coding mutations to meningomyelocele(
The contribution of de novo coding mutations to meningomyelocele( )
)
Yoo-Jin Jiny Ha et al., Nature. 2025 May;641(8062):419-426. doi: 10.1038/s41586-025-08676-x
A high-resolution transcriptomic and spatial atlas of cell types in the whole mouse brain( )
)
Zizhen Yao et al., Nature. 2023 Dec;624(7991):317-332. doi: 10.1038/s41586-023-06812-z
Molecularly defined and spatially resolved cell atlas of the whole mouse brain( )
)
Meng Zhang et al., Nature. 2023 Dec;624(7991):343-354. doi: 10.1038/s41586-023-06808-9
Single-cell DNA methylome and 3D multi-omic atlas of the adult mouse brain( )
)
Hanqing Liu et al., Nature. 2023 Dec;624(7991):366-377. doi: 10.1038/s41586-023-06805-y
Brain-wide correspondence of neuronal epigenomics and distant projections( )
)
Jingtian Zhou et al., Nature. 2023 Dec;624(7991):355-365. doi: 10.1038/s41586-023-06823-w
A transcriptomic taxonomy of mouse brain-wide spinal projecting neurons( )
)
Carla C. Winter et al.,Nature. 2023 Dec;624(7991):403-414. doi: 10.1038/s41586-023-06817-8
Spatiotemporal molecular dynamics of the developing human thalamus( )
)
Chang N Kim et al., Science. 2023 Oct 13;382(6667):eadf9941. doi: 10.1126/science.adf9941
Transcriptomic cytoarchitecture reveals principles of human neocortex organization( )
)
Nikolas L Jorstad et al., Science 2023 Oct 13;382(6667):eadf6812. doi: 10.1126/science.adf6812
Comparative transcriptomics reveals human-specific cortical features( )
)
Nikolas L Jorstad et al., Science. 2023 Oct 13;382(6667):eade9516. doi: 10.1126/science.ade9516
Single-cell analysis of prenatal and postnatal human cortical development( )
)
Dmitry Velmeshev et al., Science. 2023 Oct 13;382(6667):eadf0834. doi: 10.1126/science.adf0834
Molecular and spatial signatures of mouse brain aging at single-cell resolution( )
)
William E. Allen et al. Cell. 2023, January 5; 186, 194–208.
Modeling intercellular communication in tissues using spatial graphs of cells ( )
)
David S. Fischer, Anna C. Schaar & Fabian J. Theis. Nature Biotechnology 2022 Oct 27. doi: 10.1038/s41587-022-01467-z.
Spatially resolved epigenomic profiling of single cells in complex tissues ( )
)
Tian Lu, Cheen Euong Ang, Xiaowei Zhuang. Cell 2022 Nov 10;185(23):4448-4464.e17. doi: 10.1016/j.cell.2022.09.035. Epub 2022 Oct 21.
Pyramidal neuron subtype diversity governs microglia states in the neocortex ( )
)
Jeffrey A. Stogsdill et al., NATURE 2022
Conservation and divergence of cortical cell organization in human and mouse revealed by MERFISH ( )
)
Rongxin Fang et al., SCIENCE (2022)
Orexin neurons inhibit sleep to promote arousal ( )
)
Roberto De Luca et al., NATURE COMMUNICATIONS (2022)
Spatial Transcriptomics-correlated Electron Microscopy ( )
)
Peter Androvic et al., BIORXIV DOI:10.1101/2022.05.18.492475
Spatial transcriptomic reconstruction of the mouse olfactory glomerular map suggests principles of odor processing ( )
)
I-Hao Wang et al., NATURE NEUROSCIENCE 25, pages484–492 (2022)
Using MERSCOPE to Generate a Cell Atlas of the Mouse Brain that Includes Lowly Expressed Genes ( )
)
George Emanuel & Jiang He, MICROSCOPY TODAY (2021)
Spatially resolved cell atlas of the mouse primary motor cortex by MERFISH ( )
)
Meng Zhang et al., NATURE VOLUME 598, PAGES137–143 (2021)
GABA-receptive microglia selectively sculpt developing inhibitory circuits ( )
)
Emilia Favuzzi et al., CELL (2021)
Multi scale diffeomorphic metric mapping of spatial transcriptomics datasets ( )
)
Michael I. Miller et al., CVPR (2021)
Cell segmentation-free inference of cell types from in situ transcriptomics data ( )
)
Jeongbin Park et al., NATURE COMMUNICATIONS 12, 3545 (2021)
Spatial organization of the transcriptome in individual neurons ( )
)
Guiping Wang et al., BIORXIV DOI:10.1101/2020.12.07.414060 (2020)
Molecular, spatial and projection diversity of neurons in primary motor cortex revealed by in situ single-cell transcriptomics ( )
)
Meng Zhang et al., BIORXIV DOI:10.1101/2020.06.04.105700 (2020)
Molecular, spatial, and functional single-cell profiling of the hypothalamic preoptic region ( )
)
Jeffrey R. Moffitt et al., SCIENCE 362, 6416-EAAU5324 (2018)
High-Performance Multiplexed Fluorescence in situ Hybridization in Culture and Tissue with Matrix Imprinting and Clearing ( )
)
Jeffrey R. Moffitt et al., PNAS 113, 14456-14461 (2016)
【 がん・免疫 】
 In vivo armed macrophages curb liver metastasis through tumor-reactive T-cell rejuvenation(
In vivo armed macrophages curb liver metastasis through tumor-reactive T-cell rejuvenation( )
)
Marco Notaro et al., Nat Commun. 2025 Apr 11;16(1):3471. doi: 10.1038/s41467-025-58369-2
A multi-modal single-cell and spatial expression map of metastatic breast cancer biopsies across clinicopathological features.( )
)
Johanna Klughammer et al., Nature Medicine. 2024 Nov;30(11):3236-3249. doi: 10.1038/s41591-024-03215-z
PVRIG is Expressed on Stem-Like T Cells in Dendritic Cell-Rich Niches in Tumors and Its Blockade May Induce Immune Infiltration in Non-Inflamed Tumors.( )
)
Zoya Alteber et al., Cancer Immunol Res. 2024 Jul 2;12(7):876-890. doi: 10.1158/2326-6066.CIR-23-0752
A spatially resolved single-cell genomic atlas of the adult human breast( )
)
Tapsi Kumar et al., Nature. 2023 Jun 28. doi: 10.1038/s41586-023-06252-9
Intratumoral dendritic cell-CD4+ T helper cell niches enable CD8+ T cell differentiation following PD-1 blockade in hepatocellular carcinoma( )
)
Assaf Magen et al., Nat Med. 2023 Jun;29(6):1389-1399. doi: 10.1038/s41591-023-02345-0
Spatial analysis of human lung cancer reveals organized immune hubs enriched for stem-like CD8 T cells and associated with immunotherapy response ( )
)
Jonathan H. Chen et al., BIORXIV. 2023. DOI: https://doi.org/10.1101/2023.04.04.535379.
Intratumoral mregDC and CXCL13 T helper niches enable local differentiation of CD8 T cells following PD-1 blockade ( )
)
Assaf Magen et al., BIORXIV DOI:10.1101/2022.06.22.497216 (2022)
Interactions between cancer cells and immune cells drive transitions to mesenchymal-like states in glioblastoma ( )
)
Toshiro Hara et al., CANCER CELL (2021)
【 循環器・循環器疾患 】
 CXCL12 drives natural variation in coronary artery anatomy across diverse populations(
CXCL12 drives natural variation in coronary artery anatomy across diverse populations( )
)
Pamela E Rios Coronado et al., Cell. 2025 Apr 3;188(7):1784-1806.e22. doi: 10.1016/j.cell.2025.02.005
Single-Molecule Spatial Transcriptomics of Human Thoracic Aortic Aneurysms Uncovers Calcification-Related CARTPT-Expressing Smooth Muscle Cells( )
)
Dogukan Mizrak et al., Arterioscler Thromb Vasc Biol. 2023 Dec;43(12):2285-2297. doi: 10.1161/ATVBAHA.123.319329
【 消化器疾患 】
Charting the cellular biogeography in colitis reveals fibroblast trajectories and coordinated spatial remodeling( )
)
Paolo Cadinu et al., Cell. 2024 Apr 11;187(8):2010-2028.e30. DOI: 10.1016/j.cell.2024.03.013
【 肝臓・腎臓 】
Concordance of MERFISH spatial transcriptomics with bulk and single-cell RNA sequencing( )
)
Jonathan Liu, Vanessa Tran, et al. Life Sci Alliance. 2022 Dec 16;6(1):e202201701. doi: 10.26508/lsa.202201701.
Spatially resolved transcriptomics and the kidney: Many opportunities ( )
)
Eryn E. Dixon etr al., KIDNEY INTERNATIONAL (2022)
【 発生生物学 】
Spatial transcriptome profiling by MERFISH reveals fetal liver hematopoietic stem cell niche architecture ( )
)
Yanfang Lu et al., CELL DISCOVERY 7, 47 (2021)
Multiplexed imaging of nucleome architectures in single cells of mammalian tissue ( )
)
Miao Liu et al., NATURE COMMUNICATIONS 11, 2907 (2020)
【 植物 】
 A single-cell, spatial transcriptomic atlas of the Arabidopsis life cycle(
A single-cell, spatial transcriptomic atlas of the Arabidopsis life cycle( )
)
Travis A. Lee et al., Nat Plants. 2025 Sep;11(9):1960-1975. doi: 10.1038/s41477-025-02072-z
Time-resolved single-cell and spatial gene regulatory atlas of plants under pathogen attack( )
)
Tatsuya Nobori et al., BIORXIV. 2023. DOI: https://doi.org/10.1101/2023.04.10.536170
【 バイオインフォマティクス 】
SPEX: A modular end-to-end analytics tool for spatially resolved omics of tissues ( )
)
Ximo Pechuan Jorge et al, BIORXIV (2022)
【 方法論 】
Cell segmentation in imaging-based spatial transcriptomics ( )
)
Viktor Petukhov et al., NATURE BIOTECHNOLOGY (2021)
Genome-Scale Imaging of the 3D Organization and Transcriptional Activity of Chromatin ( )
)
Jun-Han Su et al., CELL 182(6):1641-1659.E26 (2020)
Imaging-based pooled CRISPR screening reveals regulators of lncRNA localization ( )
)
Chong Wang et al., PNAS 116 (22) 10842-10851 (2019)
Mammalian gene expression variability is explained by underlying cell state ( )
)
Robert Foreman, Roy Wollman, MOL SYST BIOL (2020)16:E9146
Spatial transcriptome profiling by MERFISH reveals subcellular RNA compartmentalization and cell cycle-dependent gene expression ( )
)
Chenglong Xia et al., PNAS 116, 19490-19499 (2019)
Multiplexed detection of RNA using MERFISH and branched DNA amplification ( )
)
Chenglong Xia et al., SCIENTIFIC REPORTS 9, 7721 (2019)
Multiplexed imaging of high-density libraries of RNAs with MERFISH and expansion microscopy ( )
)
Guiping Wang et al.,SCIENTIFIC REPORTS 8, 4847 (2018)
High-Throughput, Image-Based Screening of Genetic Variant Libraries ( )
)
George Emanuel et al., NATURE METHODS 14, 1159-1162 (2017)
High-Throughput Single-Cell Gene-Expression Profiling with Multiplexed Error-Robust Fluorescence in situ Hybridization ( )
)
Jeffrey R. Moffitt et al., PNAS 113, 11046-11051 (2016)
RNA Imaging with Multiplexed Error-Robust Fluorescence In Situ Hybridization (MERFISH) ( )
)
J.R.Moffitt, X.Zhuang, METHODS IN ENZYMOLOGY 572, 1-49 (2016)
Spatially resolved, highly multiplexed RNA profiling in single cells ( )
)
Kok Hao Chen et al., SCIENCE 348, AAA6090 (2015)
【 その他 】
Comparative analysis of multiplexed in situ gene expression profiling technologies ( )
)
Austin Hartman and Rahul Satija, eLIFE. 2024 June 7. DOI: https://doi.org/10.7554/eLife.96949.1
※この他にも多数の論文がございます。詳細はお問い合わせください。