 低品質DNAサンプル、FFPEサンプルを用いた全ゲノムシーケンス(WGS) 低品質DNAサンプル、FFPEサンプルを用いた全ゲノムシーケンス(WGS) |
全ゲノムシーケンスには、サンプルの品質がばらつく場合でも効率と一貫性が求められます。従来のワークフローでは、複数回のPCR や手動での定量が必要で、結果が不安定になりがちです。AutoNorm 機能を搭載したicon96を使えば、インプットDNA量や品質に関わらず、FFPEのようなサンプルであっても、サンプルの品質に応じた最適なサイクル数を自動的に適用し、収量のばらつきを抑えられます。その結果、1度のランでノーマライゼーション済みのライブラリーが得られ、すぐにプーリングが可能になります。
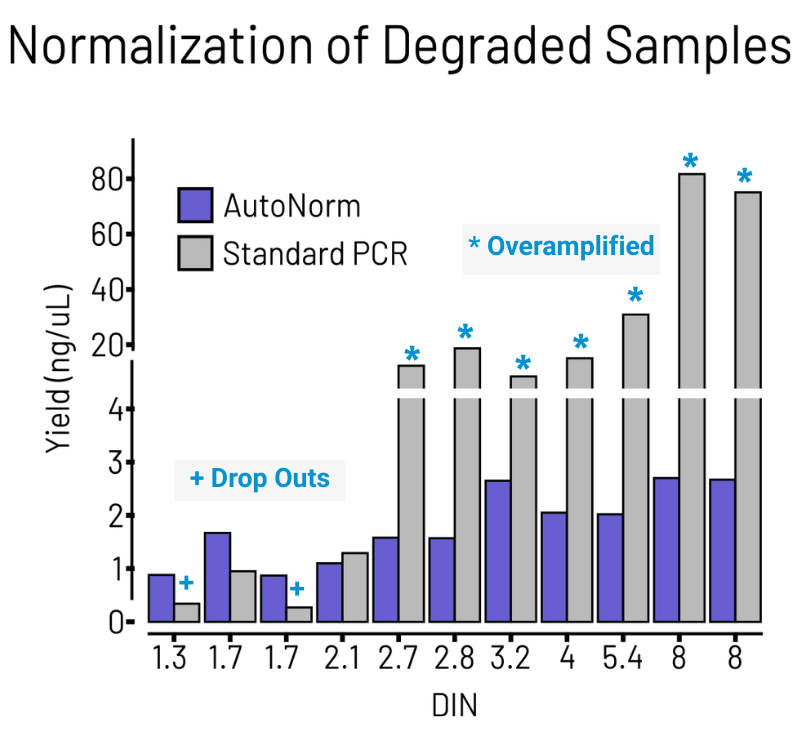
FFPEサンプルから調製したNGSライブラリーの収量。DNAの品質(DNA Integrity Number:DIN)が異なる、同等のインプット量のFFPE 由来DNAサンプルを用いてPCRを行いました。固定サイクル数を用いる従来のPCRでは、高品質なインプットDNAの多くが過剰増幅となり、低品質なインプットDNAではほとんどが収量が低く、シーケンシングに使用できませんでした(図中、StandardPCR)。一方icon96では、DNAサンプルの品質に応じてサイクル数が最適化されるため、全サンプルで収量のばらつきが抑えられ、FFPEサンプルからのNGSライブラリー調製を効率化しました(図中、AutoNorm)。
▲ アプリケーションの先頭へ戻る
 FFPEサンプルを用いたRNA-seq FFPEサンプルを用いたRNA-seq |
AutoNorm機能を搭載したicon96は、個別の精製および定量ステップを省略することでRNA-seqのライブラリー調製を効率化します。インプットRNAの品質やRNAの完全性にかかわらず、すべてのサンプルに最適な増幅サイクル数が適用され、FFPE サンプルや分解したサンプルからも信頼性の高い高品質なデータをもたらすライブラリーの生成が可能です。

AutoNorm 機能を用いて調製したライブラリーは、高品質なデータを提供します。従来のPCRまたはicon96を使用し、結腸と肝臓のFFPE サンプル(図中それぞれColon、Liver)およびコントロールサンプル(UHR+ERCC(図中UHR))から抽出した10、50、250ng のRNAからNEBNext®キットを用いてRNA-seqライブラリーを調製しました。従来のワークフローを用いて調製したライブラリーは、使用したキットが推奨する、少ないサイクル数によるPCRで調製したライブラリーと、overcycling effectを意図的に引き起こすためにサイクル数を過剰に増やして調製したライブラリーの2種類を用意しました。icon96を用いて調製したライブラリーはAutoNorm機能で増幅され、蛍光シグナルに基づいてリアルタイムでサイクル数が調整されました。
過剰なサイクル数で増幅したライブラリー(オレンジ)は、少ないサイクル数で増幅したライブラリー(濃青)やAutoNorm 機能で増幅したライブラリー(薄青)よりも、アダプターダイマーやPCR duplicate の割合が高くなり、誤ったストランドに割り当てられたリードの増加が認められました。さらに、過剰なサイクル数で増幅した結腸サンプルのライブラリーでは、Unmappedの顕著な増加が観察されました。これらの傾向は、インプット量(丸:10ng、四角:50ng、三角:250ng)にかかわらず一貫していました。このように、AutoNorm機能は過剰増幅によるアーティファクトを防ぎます。
n6 Tec社Application Note “Integrated RNA-seq Library Optimization: iconPCR™ and NEBNext® Kits Streamline Overcycling Control” より引用
▲ アプリケーションの先頭へ戻る
 FFPEサンプルを用いたDNAライブラリー調製 FFPEサンプルを用いたDNAライブラリー調製 |
FFPE由来のサンプルでは核酸の分解が進んでいるため、同一のインプット量であっても品質にばらつきが生じています。このようなサンプルを用いたDNAライブラリー調製において、従来のPCRではサイクル数が固定されていることでサイクル数が過剰もしくは過少となり、均一な増幅が達成されず、サンプルごとにPCR 前後の追加の品質管理ステップが必要となります。icon96 のAutoNorm 機能では、各サンプルの実際の収量に合わせてサイクル数を最適化することで、すべてのサンプルを均一に増幅することができます。

結腸と肝臓のFFPE サンプル(図中それぞれColon、Liver)およびコントロールサンプル(NA12878)から得られた1、10、100 ngのDNAを用いてライブラリーを調製しました。各DNAインプットは、NEBNextUltraShear® FFPE DNA Library Prep Kit(E6655L)で処理し、NEBNext® Multiplex Oligos forIllumina®(E6442S)でインデックスを付加した後、従来のPCR またはicon96で増幅しました。
従来のPCR では、1ng、10ng、100 ngのインプット量に応じて、それぞれ14、12、8サイクルのサーマルサイクラーランを個別に実施する必要がありました。一方icon96では、インプット量に関わらずすべてのサンプルを同時に処理し、各ウェルの増幅曲線の傾きが最大になった時点で増幅を自動的に終了させました(AutoNorm機能のSlope モード)。従来のPCRでは、同じ組織由来でインプット量が異なる場合や、同じインプット量でも組織の違いにより、エンドポイントのRFU値に大きなばらつきが見られました。一方icon96 では、エンドポイントのRFU 値が均一となり、一度のランにおいて、さまざまなインプット量から一貫した増幅が達成されたことが示されました。

すべてのインプット量およびサンプルタイプにおいて、固定サイクル条件とAutoNorm条件の間で品質の指標に有意な差は認められませんでした。サイクル数の違いはデータの品質に影響しないことを示しています。
n6 Tec社Application Note “Streamlined FFPE DNA Library Preparation Using NEBNext® UltraShear® and Multiplex Oligos Kits with iconPCR™” より引用
▲ アプリケーションの先頭へ戻る
 シングルセル RNA-seq シングルセル RNA-seq |
AutoNorm機能を搭載したicon96は、細胞数に関わらず最適化されたPCRサイクル数を実現します。そのため、サンプルの定量と最終的なノーマライゼーションステップを省略し、よりシンプルなワークフローで高品質のライブラリーを生成することが可能です。

従来のワークフローとicon96 によるワークフローのパフォーマンスを評価するため、GEM-X Universal 3' Gene Expression v4 チップ(10xGenomics)を用いて、シングルセル化した細胞をそれぞれ500、3000、6000、10000 細胞ずつ回収しました。cDNA 合成後、従来のワークフローもしくはicon96によるワークフローでライブラリー調製を行いました。 全サンプルから得られたデータのクラスタリングにより、共通のgene signatureが明らかになりました。このクラスタリングの結果、従来のワークフローとicon96 によるワークフローに差異は認められず、ワークフローの変更がデータ品質に影響を与えないことが示されました。
n6 Tec社 Application Note “Optimize Your Single Cell Experiments with iconPCR™” より引用
▲ アプリケーションの先頭へ戻る
 土壌サンプルのメタゲノミクス解析 土壌サンプルのメタゲノミクス解析 |
16S rRNA 遺伝子のシーケンシングは、土壌、水、ヒトに関連する微生物群など、さまざまな生態系における微生物多様性を探るために不可欠です。icon96 は多様なサンプルを自動的にノーマライズし、キメラを減少させ、プーリングを効率化します。
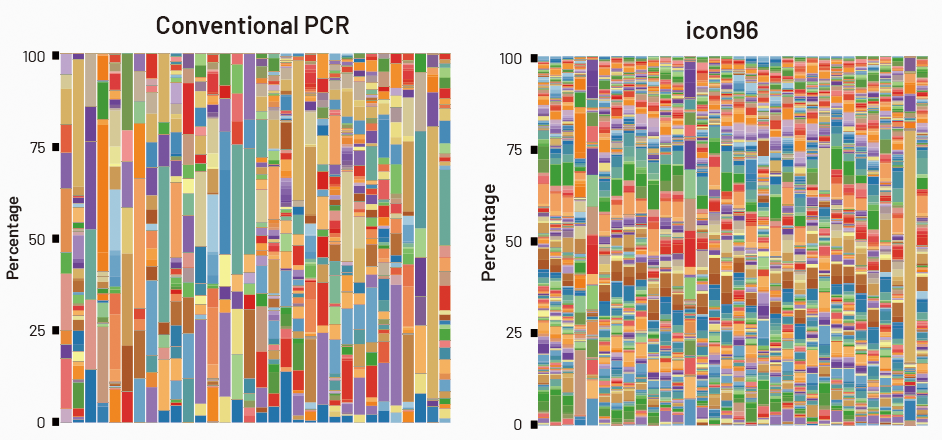
土壌サンプルのメタゲノミクス解析。メタゲノミクスのための土壌サンプルで、16S rRNA のV1-V9 領域(約1,500 bp)をカバーするロングリードシーケンスを行いました。それぞれの色は独立した細菌種を表しています。icon96を用いたワークフローにより調製されたライブラリー(icon96)は、サンプル中に豊富に含まれる細菌種の過剰増幅を防ぐことで、従来のワークフローにより調製されたライブラリー(Conventional PCR)と比べてはるかに正確な多様性レベルを示しました。

icon96を用いて調製されたライブラリー(AN)は、従来のPCR を用いて調製されたライブラリー(FC)に比べて著しく高いα多様性レベル(Chao1、Shannon、Simpson)を示しました(p < 0.001)。

β多様性プロット(Bray-Curtis 距離を使用した主座標分析(PCoA)プロット)は、icon96 を用いて調製されたライブラリー(AN)のより緊密なクラスターを示し、より高い一貫性と再現性が得られたことを示しています。
n6 Tech社Application Note “Unlocking Superior Microbial Profiling with iconPCR™” より引用
▲ アプリケーションの先頭へ戻る

(ライブ実施日:2025/10/24)
※上記画像をクリックすると別タブで視聴ページが開きます。お名前等ご入力の上、ご視聴ください。
『NGSライブラリー調製の画期的な新規ツールのご紹介』
次世代シーケンサー(NGS)を用いた解析には、全ゲノムシーケンス(WGS)、RNA-seqをはじめとする多様なアプリケーションが存在します。こうしたNGS実験の成功のカギは、ライブラリーの品質にあることが広く知られています。
特にホルマリン固定パラフィン包埋(FFPE)検体由来のDNA/RNAのように、量や品質にばらつきの大きいサンプルを扱う場合、ライブラリー調製の難易度は格段に高まります。
本セミナーでご紹介する n6 Tec社製 iconPCR™ は、これらの課題を根本的に解決する、世界初の革新的技術を搭載したサーマルサイクラーです。
特長
- ウェルごとのサーマルサイクリング個別制御
96個の独立制御可能な熱電素子を搭載し、96ウェルそれぞれを個別に温度制御できます。これにより、サンプルごとに最適なサーマルサイクリングが可能となります。
- ノーマライゼーションの全自動化
独自の AutoNorm™( オートノーマライゼーション) 機能を搭載。蛍光検出によりPCRをリアルタイムモニタリングし、最適なサイクル数で増幅を停止します。これにより、PCRと同時にノーマライゼーションが実現し、過剰増幅や過少増幅のリスクを防止します。
- ワークフローの劇的な効率化
サンプルごとに最適なサイクル数で増幅できるため、インプット量や品質の異なるサンプルを同一プレート上で同時にPCR可能です。さらに、手動でのノーマライゼーションが不要となり、プーリング後にSPRIクリーンアップを行うだけで次工程へ進めます。これらの効果により、従来のNGSワークフローと比べて最大50%の作業時間削減を実現します。
iconPCR™ を用いることで、FFPEやセルフリーDNA(cfDNA)といった低インプット/低品質サンプルからでも、アーティファクトや増幅バイアスを大幅に低減し、信頼性の高い結果を得ることが可能です。
本装置は、NGSライブラリー調製の効率化と高品質化を同時に実現できる、世界唯一のプラットフォームです。
本セミナーでは、この革新的な技術の原理から具体的な活用事例まで、わかりやすくご紹介いたします。
| 製品型式 |
icon96 |
| ウェル数 |
96 |
| サンプル容量 |
10 - 100 μL |
| 消耗品の互換性 |
96ウェルプレート(ロープロファイル)*1
8連PCRチューブ*2 |
| 加熱・冷却速度 |
0.1 - 3.0℃/秒(デフォルトは 1.5℃/s。ステップごとに設定変更可能) |
| リッド温度範囲 |
30 - 110℃ |
| 温度精度 |
±0.2℃(90℃) |
| 温度均一性 |
96ウェル全体で0.2℃ CV |
| 勾配温度制御範囲 |
4 - 98℃ |
| 蛍光検出 |
488nmレーザー、520nm蛍光(SYBR Green等対応) |
| 感度 |
単一分子検出 |
| ダイナミックレンジ |
10桁 |
| ソフトウェア |
Windows対応ソフトウェア |
| 認証 |
IEC61010-1、cTUVUS、CE、RCM |
| 動作環境 |
温度:15 - 25℃、湿度:最大80%(結露なし)、高度:海抜2,000mまで、屋内専用 |
| 汚染度 |
2 |
| IP保護等級 |
IPX0(防水性能なし) |
| 寸法 |
(W) 41 x (D) 54 x (H) 32 cm *3 |
| 重量 |
31 kg |
| 電源 |
100 – 240 V、8.5 A、50-60 Hz |
*1:メーカー検証済のプレートがございます。お問い合わせください。
*2:メーカー推奨品の8連PCRチューブを使用することも可能ですが、その場合はicon96-8連PCRチューブ用ホルダー(型式:n63002)が別途必要で、最大6列までの使用が可能です。
*3:換気のため、機器周囲に少なくとも10cmのスペースが必要です。 また、電源プラグとケーブルの容易なアクセスのため、機器背面に25~30cmのスペースが必要です。