 核染色による細胞カウントを用いたノーマライゼーション
核染色による細胞カウントを用いたノーマライゼーション異なる細胞播種密度の結果をノーマライズした実例
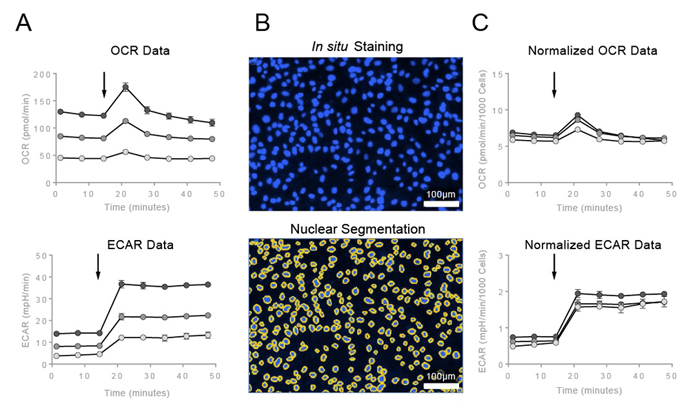
Figure 1:in situ 核染色と in situ 細胞数カウントを用いた XFデータのノーマライゼーション事例。
SKOV3細胞は 1x10
4、2x10
4、3x10
4 cells/well で播種され、24時間の培養の後、XF Cell Energy Phenotype Testが行われ、その後画像解析された。A) 20μM ヘキスト33342 (終濃度2μM) を含むオリゴマイシン+FCCP (それぞれ終濃度 1.0μM、0.5μM) の添加による OCRとECARの生データ変化。B) ヘキスト33342 によって蛍光染色された核 (上) と Cytation 1 で 認識され輪郭を描かれた核 (下) の代表的な画像。C) OCRとECARは、in situ 核染色による細胞数 (平均±SD (n=4)) によってノーマライズした。
Agilent Technologies社 Technical Overgview “Methods and strategies for normalizing XF metabolic data to cellular parameters” (5991-8980EN) より引用
ノーマライゼーションがGlycolytic Rate アッセイの結果解釈に影響を与えた実例
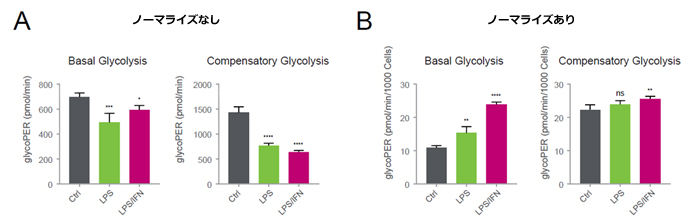
Figure 2:XF Glycolytic Rate Assayの結果に対するデータ・ノーマライゼーションの効果。
RAW264.7細胞は、100ng/ml LPS単独、または20ng/ml IFNγ と共に存在する条件にてオーバーナイトで培養し、マクロファージ活性化における解糖活性の変化をXF Glycolytic Rate Assayによって解析した。全体的なglycoPERのカイネティクスデータならびに定常状態および補償的な解糖速度を⽐較した。A) ノーマライゼーション前、B) ノーマライゼーション後。⽰されるデータは、平均±SD、n=5 technical replicates (ns: 有意でない、*: p < 0.05、**: p < 0.005、 ***:p < 0.0005、 ****:p < 0.00005)。
Agilent Technologies社 Application Note ”XF Data Normalization by the Agilent Seahorse XF Imaging and Normalization System” (5994-0022EN) より引用
 明視野画像による細胞の品質管理
明視野画像による細胞の品質管理同⼀のマイクロプレート (各ウェルに同数の細胞を含む) のウェル間におけるデータのばらつきに最も共通して⾒られる原因は、細胞分布のばらつきです。XF イメージング・ノーマライゼーションシステムは、個々のウェル画像を取得・レビューするための明視野スキャン機能を提供します。Waveソフトウェアのアッセイ結果ファイル内で個々のウェル画像をレビューし、必要に応じてフラグを⽴てることができます。
細胞播種のエラーを除くことでデータのばらつきが改善された実例
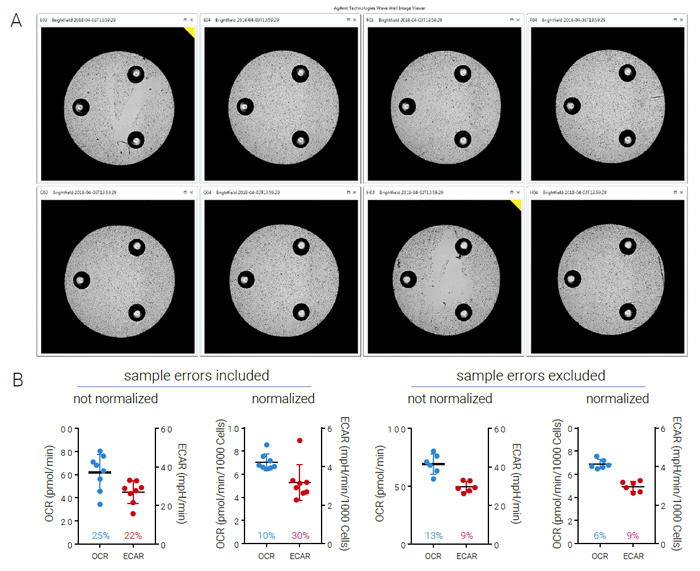
Figure 3:明視野画像を使⽤したXF解析のためのエラー同定と記録。
A) A549細胞のすべてのウェルの明視野画像をWaveソフトウェアの「ウェル比較」機能を使⽤することにより⽐較した。細胞の損失が見られるウェルは、右上角に黄色フラグで⽬印をつけている。B) 異常値除外の前後の基底OCR・ECARの⽐較。各グラフの%値 は、対応する%CVsである。
Agilent Technologies社 Technical Overview "Data Quality Management using Brightfield Images with the Seahorse XF Imaging and Normalization System" (5991-9385EN) より引用
本システムでは、BioTek Cytation 1 / BioTek Cytation 5 は新しいAgilent Seahorse XF Imaging and Cell Counting ソフトウェアによって制御されます。これによりXFアッセイデータ内で明視野画像の確認およびフラグ立て、細胞カウントによるデータのノーマライズが可能になります。
XFコントローラーのWaveソフトウェア (ver2.6) / Wave Pro ソフトウェア上で、XFアッセイデータとBioTek Cytation 1 / BioTek Cytation 5 明視野・蛍光イメージング画像を容易に統合することができます。
 |
| 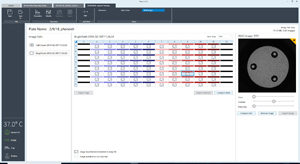 |
| 細胞数カウントデータのヒートマップ |
| エラーサンプルのフラグ立て |
|
|
|
| ウェル間における細胞数のばらつきを視覚的に表現することで、プレート内での細胞播種の均一性を容易に確認することができます。 |
| 明視野画像により、不均一な細胞分布や細胞の形態異常を示すウェルを確認することが可能です。このようなウェルは外れ値の原因となり、細胞カウントだけでは判断できない可能性があります。 |
XFイメージング・ノーマライゼーションシステムのご利用には、XFe (XFe 専用ソフトウェア ver. 2.6 以上・Wave デスクトップソフトウェア ver. 2.6 以上) / XF Pro 本体と XFe / XF Pro ノーマライゼーション機能、Agilent 社製 BioTek Cytation 1 または BioTek Cytation 5 が必要です。お持ちのコントローラーのOSにより、必要な品目が異なります。ご不明な場合はお問い合わせください。
| 製品名 | ご注文型式 |
| BioTek Cytation 1 細胞イメージングマルチモードリーダー | CYT1V-SN |
| | OBJV 4X WITH HOLDER | 1220519 |
| | CUBE LED 365 ENHANCED ASBY | 1225007 |
| | CUBE IMAGE/FILTER DAPI ASBY | 1225100 |
BioTek Cytation 5 細胞イメージングマルチモードリーダー
| |
| Seahorse XFe24 ノーマライゼーション (Win7/32bit UPG) ※1、2 | S7809B |
| Seahorse XFe96 ノーマライゼーション (Win7/32bit UPG) ※1、2 | S7809A |
| Seahorse XFe24 ノーマライゼーション (Win10/64bit 用) ※2 | S7808B |
| Seahorse XFe96 ノーマライゼーション (Win10/64bit 用) ※2 | S7808A |
| インストール・動作確認費用(XFeノーマライゼーション用UPG) | UPG_XFe |
※1:XFe コントローラー Windows 7、32bit をご使用の方は、Winsows 10、64bit へのアップ
グレードが必要です。
※2:該当機種に対応するもののみご選択ください。XFeノーマライゼーション機能 (型式:S7808A/B、S7809A/B) には、以下ハードウェア・ソフトウェアが
含まれます:
- 外付けバーコードリーダー
- Seahorse XF Imaging and Cell Counting Software Version 1.0
(XFeコントローラーにインストール)
※BioTek Cytation 1 / BioTek Cytation 5 には、BioTek Gen5ソフトウェアが付属します。
(XFeコントローラーにインストール)

Agilent社製 BioTek Cytation 1は、x4対物レンズ、DAPIフィルター、365 nm LED (DAPI検出用) 、BioTek Gen5ソフトウェアを搭載し、蛍光および高コントラスト明視野イメージングが容易に可能です。

Agilent社製 BioTek Cytation 5は、倍率1.25倍~60倍の、蛍光・明視野・ハイコントラスト明視野・カラー明視野・位相差観察 (フィルターキューブ:カラー4チャネル+明視野) が可能なイメージャーです。BioTek Gen5 ソフトウェアにより、幅広いイメージングと顕微鏡観察、およびマルチモード検出アプリケーションのワークフローを容易に自動化し、管理することができます。
※外観及び規格は予告なく変更することがございますので予めご了承ください。
※本ページに記載の製品は、すべて研究・実験用です。
人・動物の診断あるいは治療等の臨床用途に使用することはできません。